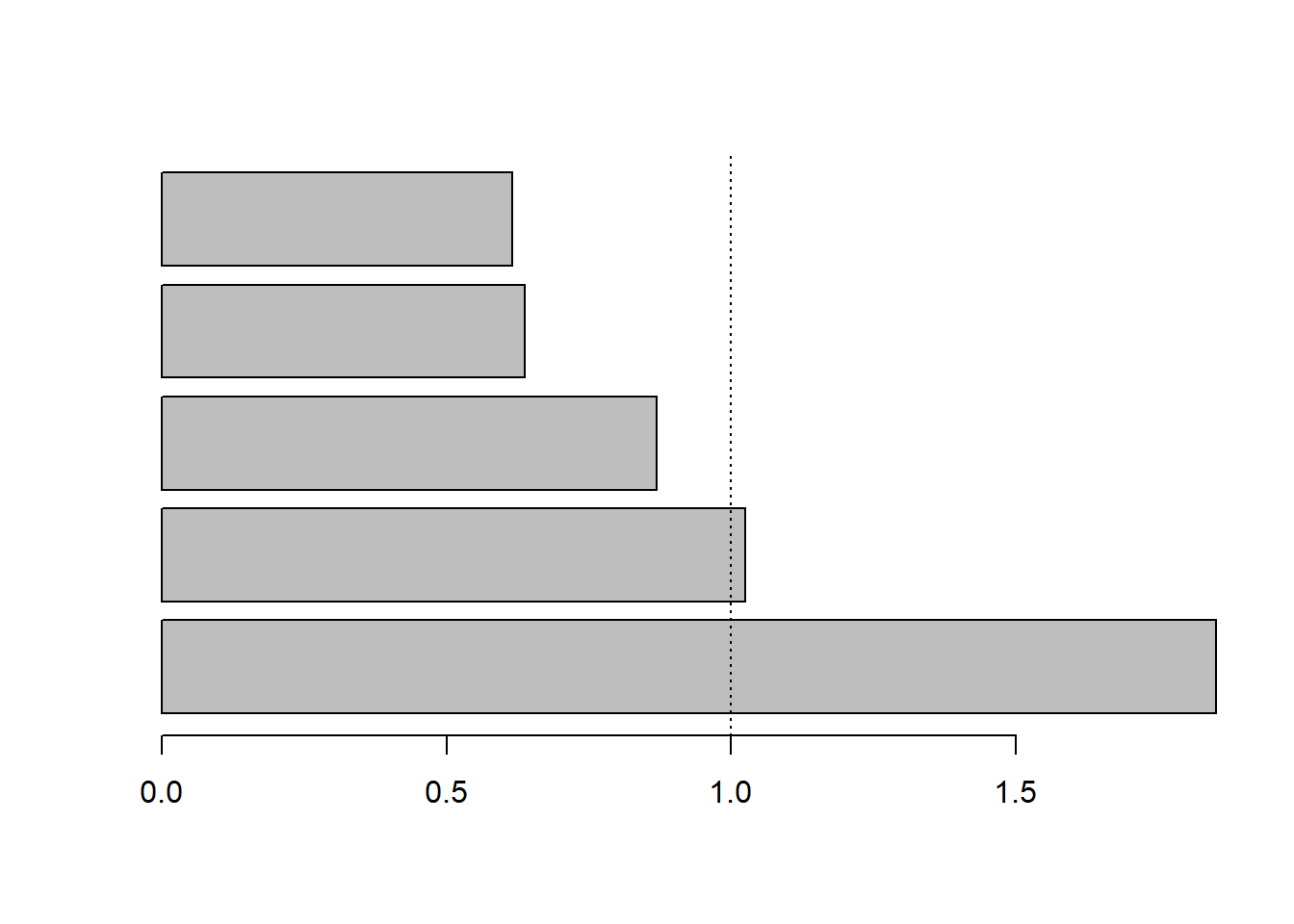2 ACP
Código a ejecutar antes de empezar:
El análisis de componentes principales (ACP) es un procedimiento estadístico que utiliza una transformación para convertir un conjunto de observaciones de variables numéricas posiblemente correlacionadas en un conjunto de valores de variables no correlacionadas linealmente llamadas componentes principales. Las componentes principales son combinación lineal (suma ponderada) de las variables originales.
El ACP permite reducir la dimensión (de manera lineal), describir relaciones lineales entre variables y comparar individuos a partir de la distancia entre ellos.
Esta transformación se define de tal manera que el primer componente principal tiene la mayor variabilidad posible (es decir, representa la mayor cantidad posible de variabilidad en los datos), y cada componente subsiguiente, a su vez, tiene la mayor variabilidad posible bajo la restricción de ser ortogonal (perpendicular) a las componentes anteriores.
A continuación utilizaremos el ejemplo que se presenta en la pág. 41 del libro. Adicionalmente, este es un extracto del resumen (abstract) del artículo origen de los datos:
“Ocasionalmente el café tostado y molido es objeto de adulteraciones, mediante la adición de toda clase de materiales extraños. Debido a que estos adulterantes modifican notablemente las características sensoriales, químicas y físicas del café, es necesario desarrollar métodos de análisis que permitan establecer el grado contenido en un producto comercial. En este estudio se analizaron 14 muestras y se realizaron determinaciones de color, acidez titulable, extracto acuoso, contenido de ácidos clorogénicos, cafeína, intensidad del aroma, acidez, amargo y cuerpo, entre otros. Se buscó sintetizar la información contenida en cada una de las determinaciones, con el fin de obtener tanto relaciones entre éstas, así como una evaluación de las muestras estudiadas. Con el análisis estadístico multivariado por componentes principales realizado se logró reducir la dimensión del problema de 16 variables estudiadas a sólo 3 factores, manteniéndose representativamente el 85,36% de la variación total…”
Duarte et al. (1996). Análisis multivariado por componentes principales, de cafés tostados y molidos adulterados con cereales. Cenicafé, 478(2):65-76
Análisis de cafés tostados y molidos adulterados con cereales
Se usa una identificación nemotécnica de los individuos, que corresponden a 2 marcas comerciales de café y a 10 tratamientos del experimento (Agregado: Sin, Maíz, Cebada. Porcentaje del Agregado: 0, 20% y 40%. Grado de tostión: Clara y Oscura).
| Color | DA | EA | pH | AcidezT | Cafeina | AcidosCl | D2325 | D2272 | Intensidad | Aroma | Cuerpo | Acidez | Amargo | Astringencia | Impresion | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ExCl | 298 | 385.1 | 25 | 5.02 | 11.7 | 1.40 | 2.74 | -0.101 | -0.057 | 7.72 | 7.00 | 6.84 | 5.02 | 5.04 | 5.36 | 7.46 |
| C40M | 361 | 481.3 | 41 | 5.11 | 6.5 | 0.81 | 1.62 | -0.058 | -0.038 | 6.02 | 5.42 | 6.22 | 4.34 | 4.60 | 4.78 | 6.24 |
| C40C | 321 | 422.6 | 40 | 5.12 | 5.8 | 0.80 | 1.61 | -0.057 | -0.038 | 6.48 | 5.98 | 6.44 | 4.58 | 4.82 | 4.80 | 6.12 |
| C20M | 335 | 444.3 | 33 | 5.05 | 8.8 | 1.10 | 2.20 | -0.079 | -0.045 | 6.82 | 6.44 | 6.70 | 4.62 | 4.38 | 4.80 | 6.04 |
| C20C | 314 | 368.7 | 32 | 5.04 | 9.3 | 1.10 | 2.19 | -0.080 | -0.046 | 7.08 | 6.20 | 6.72 | 4.78 | 4.94 | 4.90 | 6.22 |
| ExOs | 186 | 346.6 | 28 | 5.31 | 8.7 | 1.35 | 2.30 | -0.049 | -0.064 | 7.66 | 7.42 | 6.98 | 5.12 | 5.18 | 5.22 | 7.40 |
| O40M | 278 | 422.6 | 43 | 5.45 | 4.9 | 0.80 | 1.35 | -0.025 | -0.037 | 6.18 | 5.82 | 6.26 | 4.00 | 4.46 | 4.96 | 5.90 |
| O40C | 238 | 403.0 | 42 | 5.33 | 5.2 | 0.79 | 1.36 | -0.026 | -0.036 | 6.84 | 6.56 | 6.82 | 4.30 | 4.96 | 4.84 | 6.94 |
| O20M | 226 | 368.7 | 36 | 5.33 | 6.7 | 1.10 | 1.83 | -0.035 | -0.051 | 6.66 | 7.06 | 6.70 | 4.64 | 5.00 | 4.90 | 6.90 |
| O20C | 210 | 368.7 | 35 | 5.31 | 7.0 | 1.05 | 1.83 | -0.040 | -0.056 | 7.00 | 6.70 | 7.04 | 4.60 | 4.88 | 5.18 | 7.16 |
| Com1 | 221 | 413.3 | 27 | 5.18 | 9.3 | 1.30 | 2.06 | -0.053 | -0.065 | 6.48 | 5.46 | 7.06 | 4.60 | 5.26 | 5.16 | 5.68 |
| Com2 | 264 | 400.9 | 23 | 5.20 | 11.5 | 1.35 | 2.84 | -0.096 | -0.051 | 6.66 | 6.26 | 7.36 | 4.44 | 5.64 | 5.24 | 6.00 |
summary(cafe) Color DA EA pH AcidezT
Min. :186 Min. :347 Min. :23.0 Min. :5.02 Min. : 4.90
1st Qu.:225 1st Qu.:369 1st Qu.:27.8 1st Qu.:5.09 1st Qu.: 6.33
Median :271 Median :402 Median :34.0 Median :5.19 Median : 7.85
Mean :271 Mean :402 Mean :33.8 Mean :5.20 Mean : 7.95
3rd Qu.:316 3rd Qu.:423 3rd Qu.:40.2 3rd Qu.:5.32 3rd Qu.: 9.30
Max. :361 Max. :481 Max. :43.0 Max. :5.45 Max. :11.70
Cafeina AcidosCl D2325 D2272
Min. :0.790 Min. :1.35 Min. :-0.1010 Min. :-0.0650
1st Qu.:0.807 1st Qu.:1.62 1st Qu.:-0.0793 1st Qu.:-0.0563
Median :1.100 Median :1.95 Median :-0.0550 Median :-0.0485
Mean :1.079 Mean :1.99 Mean :-0.0583 Mean :-0.0487
3rd Qu.:1.312 3rd Qu.:2.23 3rd Qu.:-0.0387 3rd Qu.:-0.0380
Max. :1.400 Max. :2.84 Max. :-0.0250 Max. :-0.0360
Intensidad Aroma Cuerpo Acidez Amargo
Min. :6.02 Min. :5.42 Min. :6.22 Min. :4.00 Min. :4.38
1st Qu.:6.48 1st Qu.:5.94 1st Qu.:6.63 1st Qu.:4.42 1st Qu.:4.76
Median :6.74 Median :6.35 Median :6.77 Median :4.60 Median :4.95
Mean :6.80 Mean :6.36 Mean :6.76 Mean :4.59 Mean :4.93
3rd Qu.:7.02 3rd Qu.:6.78 3rd Qu.:7.00 3rd Qu.:4.67 3rd Qu.:5.08
Max. :7.72 Max. :7.42 Max. :7.36 Max. :5.12 Max. :5.64
Astringencia Impresion
Min. :4.78 Min. :5.68
1st Qu.:4.83 1st Qu.:6.03
Median :4.93 Median :6.23
Mean :5.01 Mean :6.50
3rd Qu.:5.19 3rd Qu.:7.00
Max. :5.36 Max. :7.46 Imagine un análisis bivariado de todas las anteriores variables (un análisis de todos los posibles pares de variables)
Para comenzar con un enfoque descriptivo multivariado, tomemos solamente los datos de dos individuos y tres variables (numéricas). Esta matriz, que denotaremos como Y, está conformada por vectores fila que representan a los individuos y por vectores columna que representan a las variables.
A partir de dicha matriz, es posible tener una representación gráfica por vectores fila (nube de individuos) y otra por vectores columna (nube de variables).
fig <- plot_ly(Aux, x = ~Color, y = ~DA, z = ~EA)
fig <- fig %>% add_markers()
fig <- fig %>% add_text(text = rownames(Aux))
fig <- fig %>% layout(title = "vectores fila\n(individuos)",
scene = list(xaxis = list(range = c(0,400)),
yaxis = list(range = c(0,400)),
zaxis = list(range = c(0,40))),
showlegend = FALSE)
figtAux <- as.data.frame(t(Aux))
fig <- plot_ly(tAux, x = ~ExCl, y = ~ExOs)
fig <- fig %>% add_markers()
fig <- fig %>% add_text(text = rownames(tAux), textposition = "top")
fig <- fig %>% layout(title = "vectores columna\n(variables)",
xaxis = list(range = c(-10,400)),
yaxis = list(range = c(-10,400)),
showlegend = FALSE)
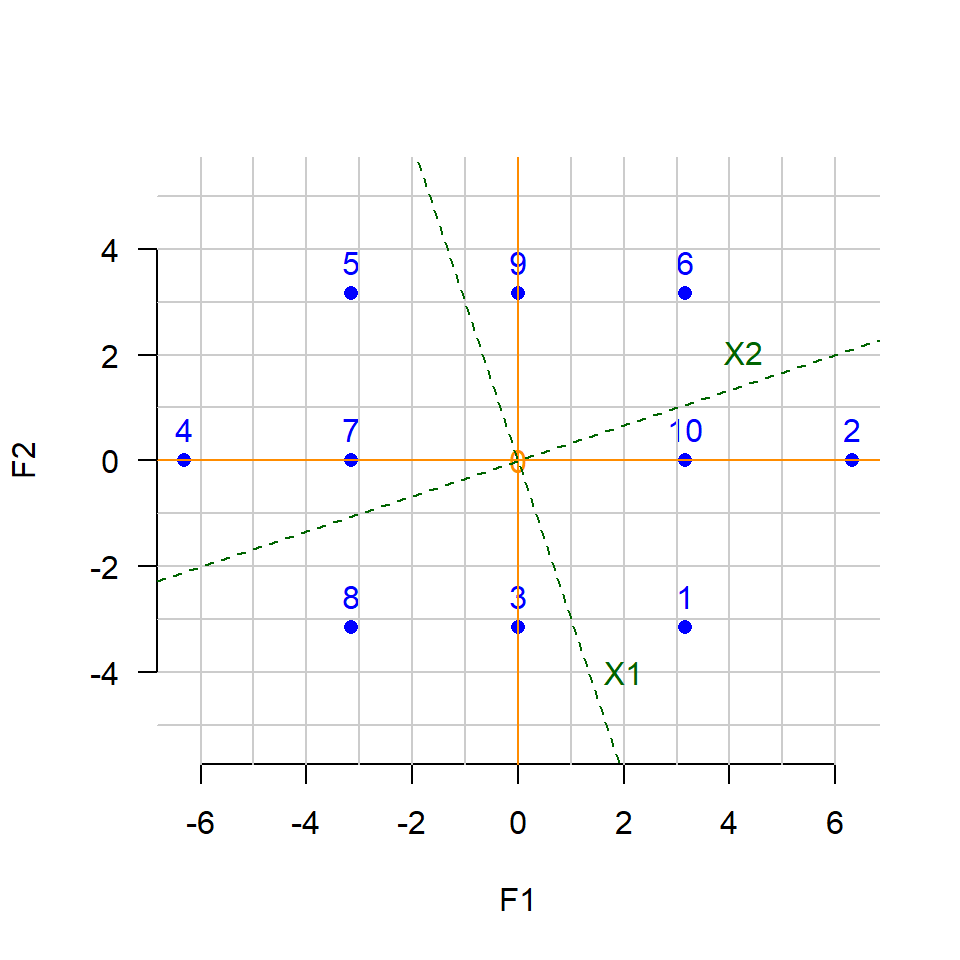
fig2.1 La nube de individuos
Cada punto corresponderá a un vector fila y representará a un individuo en un espacio de tantas dimensiones como variables.
| Color | DA | EA | |
|---|---|---|---|
| ExCl | 298 | 385.1 | 25 |
| C40M | 361 | 481.3 | 41 |
| C40C | 321 | 422.6 | 40 |
| C20M | 335 | 444.3 | 33 |
| C20C | 314 | 368.7 | 32 |
| ExOs | 186 | 346.6 | 28 |
| O40M | 278 | 422.6 | 43 |
| O40C | 238 | 403.0 | 42 |
| O20M | 226 | 368.7 | 36 |
| O20C | 210 | 368.7 | 35 |
2.1.1 Centro de gravedad
Es una suma ponderada de los vectores fila (uno vector por individuo). \vec{\mathrm{g}} = \sum_{i = 1}^n p_i \vec{y}_i
Cuando todos los pesos p_i son iguales a \frac{1}{n} (o lo que es lo mismo, cuando todos los individuos tienen el mismo peso o ponderación), es la suma de los vectores fila, para luego dividir por la cantidad de individuos, \begin{aligned} \vec{\mathrm{g}} &= \frac{\vec{y}_1 + \vec{y}_2 + \dots + \vec{y}_n}{n} \\ &= \frac{1}{n} \vec{y}_1 + \frac{1}{n} \vec{y}_2 + \dots + \frac{1}{n} \vec{y}_n \\ &= \sum_{i = 1}^n \frac{1}{n} \vec{y}_i \end{aligned}
par(las = 1) # gráfica
Y3D <- scatterplot3d(Y, main = "Y", type = "h", color = 4,
box = FALSE, las = 1)
Y3D$points3d(Y, pch = 16, col = 4)
addgrids3d(Y, grid = c("xy","xz","yz"))
cord2d <- Y3D$xyz.convert(Y) # convertir coordenadas 3D a 2D
# poner etiquetas:
text(cord2d, labels = rownames(Y), cex = 0.8, pos = 3)
g <- colMeans(Y) # centro de gravedad
Y3D$points3d(t(g), pch = 19, col = 3, type = "h")
text(Y3D$xyz.convert(t(g)), labels = "g",
pos = 3, cex = 1.3)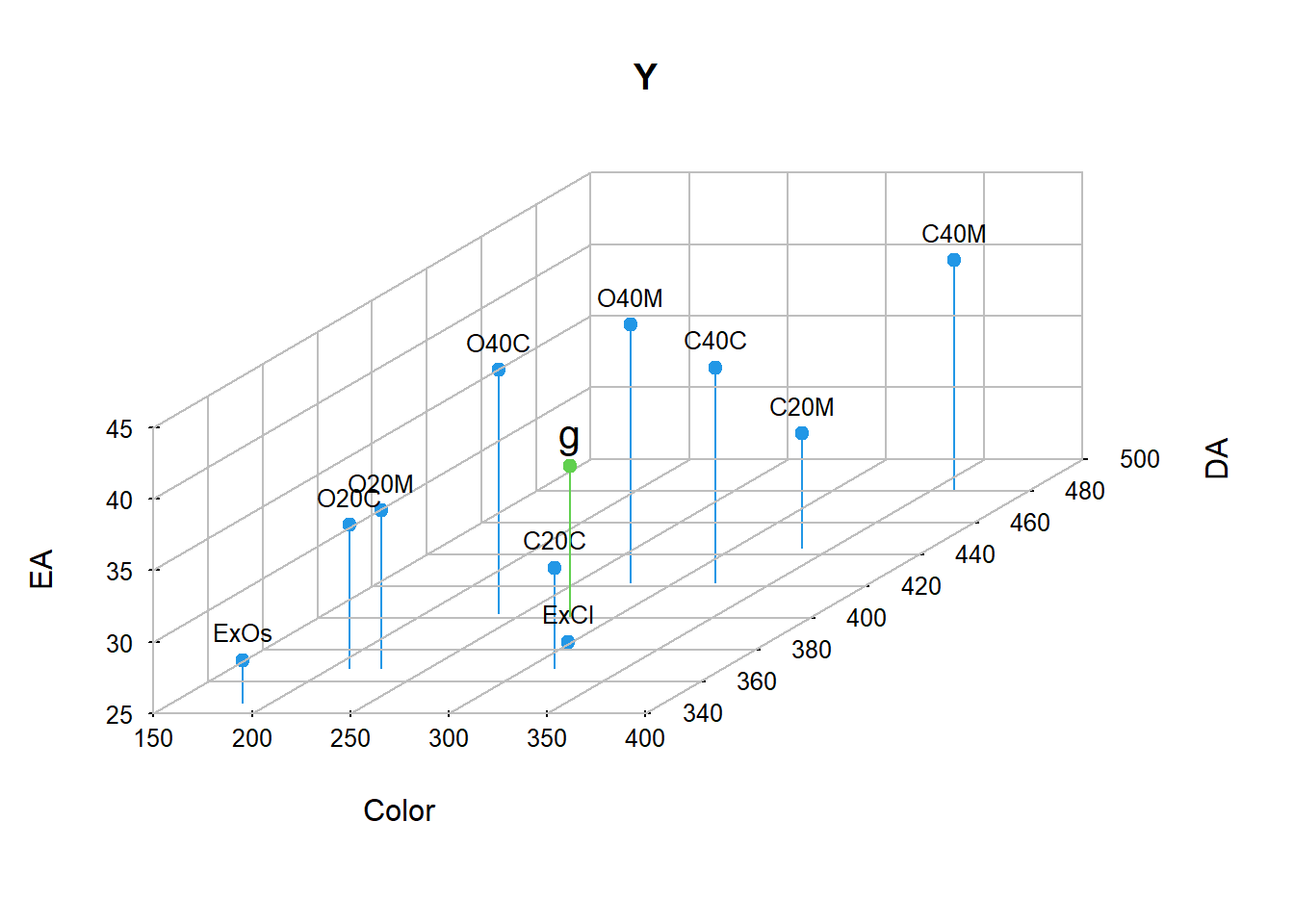
2.1.2 Centrado
Centrar los puntos es hacer que el centro de gravedad quede en el origen de las coordenadas (“en el cero”).
A cada vector fila se le resta el vector de medias, de esta manera se da una traslación de todos los individuos. Esta operación se escribiría así para cada vector fila (para el i-ésimo): \vec{y}_{Ci} = \vec{y}_i - \vec{\mathrm{g}} y matricialmente para todos los individuos a la vez, así: Y_C = Y - \mathbf{1}_n \, \vec{\mathrm{g}}'
| Color | DA | EA | |
|---|---|---|---|
| ExCl | 21.3 | -16.06 | -10.5 |
| C40M | 84.3 | 80.14 | 5.5 |
| C40C | 44.3 | 21.44 | 4.5 |
| C20M | 58.3 | 43.14 | -2.5 |
| C20C | 37.3 | -32.46 | -3.5 |
| ExOs | -90.7 | -54.56 | -7.5 |
| O40M | 1.3 | 21.44 | 7.5 |
| O40C | -38.7 | 1.84 | 6.5 |
| O20M | -50.7 | -32.46 | 0.5 |
| O20C | -66.7 | -32.46 | -0.5 |
# grafica de datos centrados
Yc3D <- scatterplot3d(Yc, main = "Yc", type = "h",
color = 4, box = FALSE, las = 1)
Yc3D$points3d(Yc, pch = 16, col = 4)
addgrids3d(Yc, grid = c("xy","xz","yz"))
text(Yc3D$xyz.convert(Yc), labels = rownames(Yc),
cex = 0.8, pos = 3)
Yc3D$points3d(t(c(0,0,0)), pch = 19, col = 3, type = "h")
text(Yc3D$xyz.convert(t(c(0,0,0))), labels = "0",
pos = 3, cex = 1.3)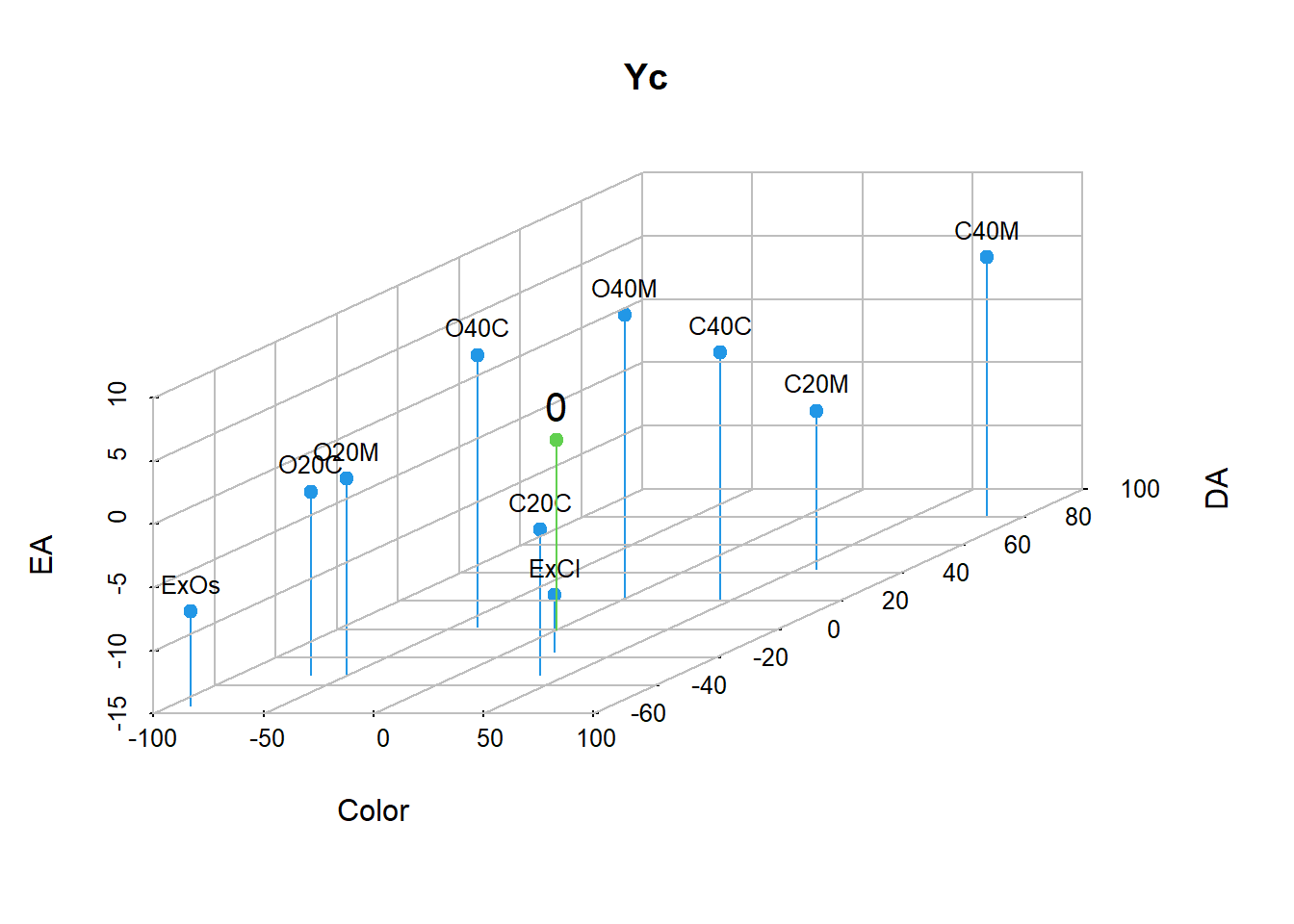
2.1.3 Distancia entre individuos
fig <- plot_ly(Yc, x = ~Color, y = ~DA, z = ~EA)
fig <- fig %>% add_markers()
fig <- fig %>% add_text(text = rownames(Yc))
fig <- fig %>% add_markers(x = 0, y = 0, z = 0)
fig <- fig %>% layout(showlegend = FALSE)
figLa distancia euclidiana al cuadrado entre dos individuos (dos vectores fila) es la suma de las diferencias al cuadrado de sus componentes variable por variable: d^2(\vec{y}_i,\vec{y}_l) = \sum_{j=1}^p ( y_{ij} - y_{lj} )^2 Suma ponderada en donde cada diferencia al cuadrado (una por cada variable) tiene la misma ponderación: 1.
ExCl C40M C40C C20M C20C ExOs O40M O40C O20M
C40M 116.1
C40C 46.5 71.0
C20M 70.3 45.9 26.8
C20C 24.0 122.3 54.9 78.5
ExOs 118.5 221.2 155.4 178.2 130.0
O40M 46.2 101.7 43.1 61.8 65.7 120.3
O40C 64.9 145.8 85.3 105.8 84.0 78.0 44.6
O20M 74.7 175.9 109.3 132.7 88.1 46.4 75.2 36.8
O20C 90.1 188.5 123.5 146.1 104.0 33.4 87.1 44.8 16.02.1.4 Inercia
La inercia de la nube de individuos es, \begin{aligned} Inercia(N_n) &= \sum\limits_{i=1}^{n}{p_i \, d^2\left(\vec{y}_i,\vec{\mathbf{g}}\right)} \\ &= \sum\limits_{i=1}^{n}{p_i \, d^2\left(\vec{y}_{Ci} ,\vec{0}\right)} \\ &= \sum\limits_{i=1}^{n}{p_i \, \left(\sum\limits_{j=1}^{p}y_{{_C}_{ij}}^2\right)} \end{aligned} Cuando todos los pesos p_i son iguales a \frac{1}{n}, \begin{aligned} Inercia(N_n) &= \dfrac{1}{n}\sum\limits_{i=1}^{n}\sum\limits_{j=1}^{p}y_{{_C}_{ij}}^2 \\ &= \sum\limits_{j=1}^{p}\dfrac{1}{n}\sum\limits_{i=1}^{n}y_{{_C}_{ij}}^2 \\ &= \sum\limits_{j=1}^{p}\sigma_j^2 \end{aligned}
Concluimos que la inercia de la nube de individuos es la suma de las varianzas de las variables.
La matriz de varianzas y covarianzas de \mathbf{Y} (que es la misma matriz de varianzas y covarianzas de \mathbf{Y}_C) es, V=\dfrac{1}{n} Y'_C Y_C La suma de las varianzas, es decir la inercia de la nube de individuos, es igual a traza(V).
2.1.5 Reducción
Si a cada término de la matriz Y lo centramos y lo reducimos (lo estandarizamos), entonces obtenemos, x_{ij}=\dfrac{y_{ij}-\bar{y}_j}{\sigma_j} donde \bar{y}_j y \sigma_j son respectivamente la media y la desviación estándar de la variable j.
Matricialmente, X = Y_C \, D_{\sigma}^{-1} donde D_{\sigma}=diag(\sigma_{_j}).
var(Y)*(n-1)/n Color DA EA
Color 3105.81 1738.4 60.95
DA 1738.39 1560.2 129.36
EA 60.95 129.4 33.45 Color DA EA
Color 3105.81 1738.4 60.95
DA 1738.39 1560.2 129.36
EA 60.95 129.4 33.45| Color | DA | EA | |
|---|---|---|---|
| ExCl | 0.4 | -0.4 | -1.8 |
| C40M | 1.5 | 2.0 | 1.0 |
| C40C | 0.8 | 0.5 | 0.8 |
| C20M | 1.0 | 1.1 | -0.4 |
| C20C | 0.7 | -0.8 | -0.6 |
| ExOs | -1.6 | -1.4 | -1.3 |
| O40M | 0.0 | 0.5 | 1.3 |
| O40C | -0.7 | 0.0 | 1.1 |
| O20M | -0.9 | -0.8 | 0.1 |
| O20C | -1.2 | -0.8 | -0.1 |
par(las = 1) # etiquetas en los dos ejes serán horizontales
X3D <- scatterplot3d(X, main = "X", type = "h",
box = FALSE, color = 4, asp = 1)
X3D$points3d(X, pch = 16, col = 4)
addgrids3d(X, grid = c("xy","xz","yz"))
text(X3D$xyz.convert(X), labels = rownames(X), cex =0.8, pos =3)
X3D$points3d(t(c(0,0,0)), pch = 19, col = 3, type = "h")
text(X3D$xyz.convert(t(c(0,0,0))), labels = "0",
pos = 3, cex = 1.3)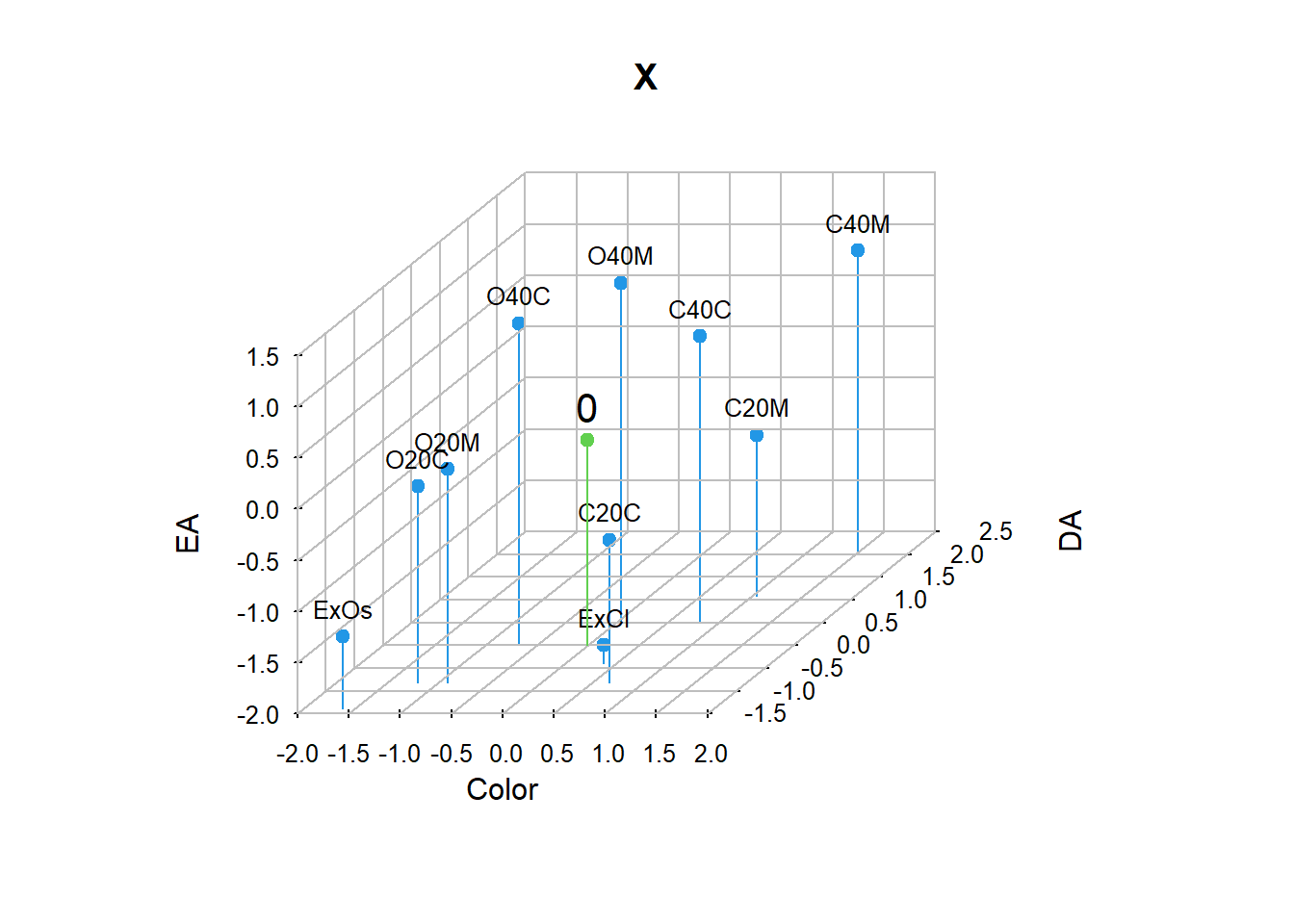
V <- var(Y)*(n-1)/n # Varianza poblacional
Dsigma <- diag(sqrt(diag(V))) # Desv. estandar
X <- as.matrix(Yc) %*% solve(Dsigma)
colnames(X) <- colnames(Y)
X <- as.data.frame(X)
fig <- plot_ly(X, x = ~Color, y = ~DA, z = ~EA)
fig <- fig %>% add_markers(text = rownames(X))
fig <- fig %>% add_text(text = rownames(X))
fig <- fig %>% add_markers(x = 0, y = 0, z = 0)
fig <- fig %>% layout(showlegend = FALSE)
figfig <- plot_ly(X, x = ~Color, y = ~DA)
fig <- fig %>% add_markers(text = rownames(X))
fig <- fig %>% add_text(text = rownames(X), textposition = "top")
fig <- config(fig, displayModeBar = FALSE) %>%
layout(xaxis = list(range = c(-2.5,2.5)),
yaxis = list(range = c(-2.5,2.5)),
showlegend = FALSE)
figfig <- plot_ly(X, x = ~Color, y = ~EA)
fig <- fig %>% add_markers(text = rownames(X))
fig <- fig %>% add_text(text = rownames(X), textposition = "top")
fig <- config(fig, displayModeBar = FALSE) %>%
layout(xaxis = list(range = c(-2.5,2.5)),
yaxis = list(range = c(-2.5,2.5)),
showlegend = FALSE)
figfig <- plot_ly(X, x = ~DA, y = ~EA)
fig <- fig %>% add_markers(text = rownames(X))
fig <- fig %>% add_text(text = rownames(X), textposition = "top")
fig <- config(fig, displayModeBar = FALSE) %>%
layout(xaxis = list(range = c(-2.5,2.5)),
yaxis = list(range = c(-2.5,2.5)),
showlegend = FALSE)
fig ExCl C40M C40C C20M C20C ExOs O40M O40C O20M
C40M 3.9
C40C 2.8 1.7
C20M 2.1 1.7 1.4
C20C 1.3 3.4 1.9 2.0
ExOs 2.3 5.2 3.7 3.7 2.5
O40M 3.3 2.1 0.9 2.1 2.4 3.6
O40C 3.2 3.0 1.6 2.6 2.4 3.0 0.9
O20M 2.3 3.8 2.3 2.8 1.7 1.7 2.0 1.4
O20C 2.4 4.1 2.6 3.0 1.9 1.4 2.3 1.6 0.3La matriz de varianzas y covarianzas de X (que es la misma matriz de correlaciones de \mathbf{Y}_C y de \mathbf{Y}) es, V_X= \dfrac{1}{n} X'X
var(X)*(n-1)/n Color DA EA
Color 1.0000 0.7897 0.1891
DA 0.7897 1.0000 0.5663
EA 0.1891 0.5663 1.0000 Color DA EA
Color 1.0000 0.7897 0.1891
DA 0.7897 1.0000 0.5663
EA 0.1891 0.5663 1.0000cor(Y) Color DA EA
Color 1.0000 0.7897 0.1891
DA 0.7897 1.0000 0.5663
EA 0.1891 0.5663 1.0000Además, la inercia de la nube de individuos con sus coordenadas estandarizadas es, \begin{aligned} Inercia\left(N_{n_X}\right) &= 1 + 1 + \dots + 1 = p \end{aligned}
2.1.6 Nuevos ejes (cambio de base)
“A function \left\langle \,\cdot ,\cdot \,\right\rangle :\mathbb{R}^{p}\times \mathbb{R}^{p}\to \mathbb{R} is an inner product on \mathbb{R}^{p} if and only if there exists a symmetric positive-definite matrix \mathbf{M} such that \left\langle u,v\right\rangle =u' \mathbf {M} v for all u,v \in \mathbb {R} ^{p}. If \mathbf{M} is the identity matrix then \left\langle u,v\right\rangle =u' \mathbf{M} v = u' v is the dot product.”
Norma:
\left\| u \right\|^2 = \left\langle u,u\right\rangle
Ángulo:
\begin{aligned} \cos\left(\angle_{\vec{u}}^{\vec{v}}\right) &= \frac{\left\langle u,v \right\rangle}{\left\| u \right\| \left\| v \right\|} \end{aligned} de donde se tiene que, \left\langle u,v \right\rangle = \cos\left(\angle_{\vec{u}}^{\vec{v}}\right) \left\| u \right\| \left\| v \right\|
La proyección ortogonal de un vector x_i (individuo i-ésimo) sobre un vector unitario u (\left\| u \right\| = 1) es, \begin{aligned} &\left\| x_i \right\| \cos\left(\angle_{\vec{u}}^{\vec{v}}\right) \\ &= \left\| x_i \right\| \frac{\left\langle x_i,u \right\rangle}{\left\| x_i \right\| \left\| u \right\|} \\ &= \left\langle x_i,u \right\rangle \end{aligned}
La inercia de la nube de individuos sobre la recta que genera el vector u es, \begin{aligned} \sum_{i = 1}^{n} p_i \, d^2\left(\left\langle x_i,u \right\rangle, 0\right) &= \frac{1}{n} \sum_{i = 1}^{n} \left(\left\langle x_i,u \right\rangle\right)^2 \\ &= \frac{1}{n} \sum_{i = 1}^{n} \left( x_i' u \right)^2 \\ &= \frac{1}{n} \left(Xu\right)' \left(Xu\right) \\ &= \frac{1}{n} u'X'Xu \\ &= u' \left( \frac{1}{n} X'X \right) u \\ &= u' \, V_X \, u \end{aligned}
¿Cuál sería el vector unitario que hace que la inercia de la nube sobre la recta que genera el vector sea la máxima posible?
\begin{aligned} {\underset {u \in \mathbb{R}^p}{\text{arg max}}}\quad & u' \left( \frac{1}{n} X'X \right) u \\ {\text{subject to}}\quad &u'u - 1 = 0. \end{aligned}
Usando multiplicadores de Lagrange, f(u) = u' \left( \frac{1}{n} X'X \right) u - \lambda \left( u'u - 1 \right) que tiene como primera derivada, \frac{d}{du} f(u) = 2 \left( \frac{1}{n} X'X \right) u - 2 \lambda u Igualando a cero, \begin{aligned} 2 \left( \frac{1}{n} X'X \right) u - 2 \lambda u &= 0 \\ \left( \frac{1}{n} X'X \right) u &= \lambda u \\ V_X \, u &= \lambda u \end{aligned}
u tiene que ser un vector propio y \lambda tiene que ser un valor propio (todos son candidatos a ser el que maximiza). ¿Pero cuál de todos ellos maximiza la inercia u' \left( \frac{1}{n} X'X \right) u?
\begin{aligned} \left( \frac{1}{n} X'X \right) u &= \lambda u \\ u' \left( \frac{1}{n} X'X \right) u &= u'\lambda u \\ u' \left( \frac{1}{n} X'X \right) u &= \lambda \\ u' \, V_X \, u = \lambda \end{aligned}
De todos los vectores candidatos, el vector propio (unitario) asociado al valor propio más grande, es el que genera la recta sobre la cual la inercia de la nube es máxima.
Sea \lambda_1 el valor propio más grande y u_1 su respectivo vector propio unitario. El vector -u_1 también es solución del problema de optimización planteado. El significado del sentido del eje se busca a partir de las variables, ya que el signo de las coordenadas depende del vector propio seleccionado.
Las coordenadas de los individuos sobre el eje generado por u_1, denominado primer eje principal, se denotan por F_1 y son: F_1 = X u_1
Ya se obtuvo la recta sobre la cual la inercia de la nube es máxima. Para obtener el plano sobre el cual la inercia de la nube es máxima, se busca una segunda recta o eje generado por un vector unitario ortogonal a u_1 que tenga la mayor inercia posible de la nube de individuos. Es decir,
\begin{aligned} {\underset {u \in \mathbb{R}^p}{\text{arg max}}}\quad & u' \left( \frac{1}{n} X'X \right) u \\ {\text{subject to}}\quad &u'u - 1 = 0. \\ & u'u_1 = 0 \end{aligned}
Usando multiplicadores de Lagrange, f(u) = u' \left( \frac{1}{n} X'X \right) u - \lambda \left( u'u - 1 \right) - \mu \left(u'u_1\right) Que tiene como primera derivada, \frac{d}{du} f(u) = 2 \left( \frac{1}{n} X'X \right) u - 2 \lambda u - \mu u_1 Igualando a cero, \begin{aligned} 2 \left( \frac{1}{n} X'X \right) u - 2 \lambda u - \mu u_1 &= 0 \\ 2 u_1' \left( \frac{1}{n} X'X \right) u - 2 \lambda u_1' u - \mu u_1' u_1 &= 0 \\ \mu &= 0 \end{aligned} de donde nos queda que, \begin{aligned} 2 \left( \frac{1}{n} X'X \right) u - 2 \lambda u &= 0 \\ \left( \frac{1}{n} X'X \right) u &= \lambda u \\ V_X \, u &= \lambda u \end{aligned} De nuevo, todos los vectores propios son candidatos. Pero el que maximiza la inercia es aquel con mayor valor propio, que además debe ser ortogonal al primer valor propio, es decir, u tendría que ser el segundo vector propio unitario (o su inverso aditivo).
Siguiendo la misma lógica, encontramos que el subespacio 3D en donde la inercia de la nube es máxima sería el generado por los tres vectores propios unitarios asociados a los tres valores propios más grandes de \frac{1}{n} X'X = V_X.
El número de valores propios diferentes de \frac{1}{n} X'X coincide con el rango de \frac{1}{n} X'X, que si n > p entonces coincide con el número de vectores columna (variables) linealmente independientes de X.
Si r = p, los p vectores propios constituyen una base ortonormal para el espacio de la nube de individuos, que permite, en orden, las proyecciones de mayor inercia.
Las n coordenadas de los individuos sobre un eje factorial s, F_s, constituyen los valores de una variable nueva denominada componente principal, F_s = X u_s Su varianza es, \begin{aligned} \frac{1}{n}\sum_{i=1}^n \left[F_s(i)\right]^2 &= \frac{1}{n}F'_s F_s \\ &= \frac{1}{n} \left( X u_s \right)' X u_s \\ &= u'_s \left( \frac{1}{n} X'X \right) u_s \\ &= \lambda_s \end{aligned}
Note que (si r = p) la inercia de la nube de individuos estandarizada (centrada y reducida) es, traza\left(\frac{1}{n} X'X\right) = p = \sum_{s=1}^p \lambda_s
# V = t(X) %*% as.matrix(X)/n = var(X)*(n-1)/n = cor(Y)
des <- eigen(V)
lambda <- des$values
names(lambda) <- paste("lambda_", 1:p)
res <- data.frame(var = lambda, perc = 100*lambda/p)
res <- cbind(res, acum = cumsum(res$perc))
res <- rbind(res, Total = c(colSums(res[,1:2]), NA))
kable(res, digits = 2)| var | perc | acum | |
|---|---|---|---|
| lambda_ 1 | 2.07 | 68.90 | 68.90 |
| lambda_ 2 | 0.82 | 27.39 | 96.29 |
| lambda_ 3 | 0.11 | 3.71 | 100.00 |
| Total | 3.00 | 100.00 | NA |
U <- des$vectors
rownames(U) <- rownames(V)
colnames(U) <- c("Eje1","Eje2","Eje3")
kable(as.data.frame(U), digits = 3)| Eje1 | Eje2 | Eje3 | |
|---|---|---|---|
| Color | 0.579 | -0.571 | 0.581 |
| DA | 0.673 | -0.067 | -0.737 |
| EA | 0.460 | 0.818 | 0.346 |
Las coordenadas de la nube de individuos en los nuevos ejes (en las componentes principales) se pueden obtener haciendo, F = X U en donde los vectores propios (ordenados por su valor propio correspondiente) son los vectores columna de la matriz U.
F <- as.matrix(X) %*% U
F_df <- as.data.frame(F)
kable(F_df, digits = 3)| Eje1 | Eje2 | Eje3 | |
|---|---|---|---|
| ExCl | -0.887 | -1.676 | -0.106 |
| C40M | 2.679 | -0.222 | -0.287 |
| C40C | 1.184 | 0.146 | 0.331 |
| C20M | 1.142 | -1.024 | -0.346 |
| C20C | -0.443 | -0.823 | 0.785 |
| ExOs | -2.469 | -0.038 | -0.377 |
| O40M | 0.975 | 1.011 | 0.062 |
| O40C | 0.146 | 1.313 | -0.049 |
| O20M | -1.040 | 0.645 | 0.107 |
| O20C | -1.286 | 0.668 | -0.120 |
Teniendo en cuenta todo lo anterior, ¿hasta ahora, para qué podríamos llegar a utilizar un análisis de componentes principales? (¿que utilidad le puedo encontrar a todo el proceso realizado con la nube de individuos?)
tpos <- rep(c("top","bottom"), length.out = n)[rank(F[,1])]
fig <- plot_ly(F_df, x = ~Eje1, y = "")
fig <- fig %>% add_markers(marker = list(symbol = 4),
hovertext = rownames(F_df))
fig <- fig %>% add_text(text = rownames(F_df),
textposition = tpos)
fig <- fig %>% layout(showlegend = FALSE)
figfig <- plot_ly(F_df, x = ~Eje1, y = ~Eje2)
fig <- fig %>% add_markers(hovertext = rownames(F_df))
fig <- fig %>% add_text(text = rownames(F_df), textposition = tpos)
fig <- fig %>% layout(showlegend = FALSE)
figfig <- plot_ly(F_df, x = ~Eje1, y = ~Eje2, z = ~Eje3)
fig <- fig %>% add_markers()
fig <- fig %>% add_text(text = rownames(F_df))
fig <- fig %>% add_markers(x = 0, y = 0, z = 0)
fig <- fig %>% layout(showlegend = FALSE)
fig2.1.7 Ayudas interpretación
Distancia al origen:
\begin{aligned} d^2\left(\vec{x}_i, 0\right) &= \left\|\vec{x}_i\right\|^2 \\ &= \sum_{j=1}^p x_{ij}^2 \\ &= \sum_{j=1}^p \left[ \left\langle \vec{x}_i, \vec{e}_j \right\rangle \right]^2 \\ &= \sum_{s=1}^p \left[ \left\langle \vec{x}_i, \vec{u}_s \right\rangle \right]^2 \\ &= \sum_{s=1}^p \left[ \vec{x}_i' \vec{u}_s \right]^2 \\ &= \sum_{s=1}^p \left[F_s(i)\right]^2 \end{aligned}
Contribución absoluta:
\begin{aligned} Inercia_s (N_n) = \sum_{i=1}^n p_i \left[F_s(i)\right]^2 &= \lambda_s \\ \sum_{i=1}^n \frac{p_i \left[F_s(i)\right]^2}{\lambda_s} &= 1 \\ \sum_{i=1}^n Ca_s(i) = 1 \end{aligned} donde, Ca_s(i) = \frac{p_i \left[F_s(i)\right]^2}{\lambda_s} con p_i = \frac{1}{n} cuando todos los individuos tienen el mismo peso.
Coseno cuadrado:
\begin{aligned} \cos \left(\angle_s^{\vec{x}_i}\right) &= \frac{\left\langle \vec{x}_i, \vec{u}_s \right\rangle}{\left\| \vec{x}_i \right\| \left\| \vec{u}_s \right\|} \in [-1,1] \end{aligned}
\begin{aligned} \cos^2 \left(\angle_s^{\vec{x}_i}\right) &= \frac{\left[ \left\langle \vec{x}_i, \vec{u}_s \right\rangle \right]^2}{\left\| \vec{x}_i \right\|^2} \in [0,1] \\ &= \frac{\left[F_s(i)\right]^2}{\left\| \vec{x}_i \right\|^2} \end{aligned}
\begin{aligned} \sum_{s=1}^p \cos^2 \left(\angle_s^{\vec{x}_i}\right) &= \frac{\sum_{s=1}^p \left[F_s(i)\right]^2}{\left\| \vec{x}_i \right\|^2} \\ &= \frac{\left\|\vec{x}_i\right\|^2}{\left\| \vec{x}_i \right\|^2} \\ &= 1 \end{aligned}
# tabla de ayudas para la interpretacion
# sobre el primer plano factorial
d2 <- rowSums(F^2) # distancias
cont <- 1/n*F^2 %*% diag(1/lambda)*100 # contribuciones
cos2 <- F^2/d2*100 # cosenos cuadrados
Ayu <- cbind(dis2 = d2, F1 = F[,1], F2=F[,2],
cont1 = cont[,1], cont2 = cont[,2],
cos21 = cos2[,1], cos22 = cos2[,2],
cosp = rowSums(cos2[,1:2]))
Ayu <- rbind(Ayu, Total = c(colSums(Ayu[,1:5]), rep(NA, 3)))
kable(Ayu, digits = 2)| dis2 | F1 | F2 | cont1 | cont2 | cos21 | cos22 | cosp | |
|---|---|---|---|---|---|---|---|---|
| ExCl | 3.61 | -0.89 | -1.68 | 3.80 | 34.19 | 21.80 | 77.89 | 99.69 |
| C40M | 7.31 | 2.68 | -0.22 | 34.72 | 0.60 | 98.20 | 0.67 | 98.87 |
| C40C | 1.53 | 1.18 | 0.15 | 6.78 | 0.26 | 91.45 | 1.39 | 92.84 |
| C20M | 2.47 | 1.14 | -1.02 | 6.31 | 12.77 | 52.75 | 42.41 | 95.16 |
| C20C | 1.49 | -0.44 | -0.82 | 0.95 | 8.23 | 13.20 | 45.42 | 58.62 |
| ExOs | 6.24 | -2.47 | -0.04 | 29.49 | 0.02 | 97.70 | 0.02 | 97.73 |
| O40M | 1.98 | 0.98 | 1.01 | 4.60 | 12.44 | 48.09 | 51.72 | 99.81 |
| O40C | 1.75 | 0.15 | 1.31 | 0.10 | 20.98 | 1.21 | 98.65 | 99.86 |
| O20M | 1.51 | -1.04 | 0.65 | 5.24 | 5.07 | 71.67 | 27.58 | 99.25 |
| O20C | 2.12 | -1.29 | 0.67 | 8.00 | 5.43 | 78.22 | 21.10 | 99.32 |
| Total | 30.00 | 0.00 | 0.00 | 100.00 | 100.00 | NA | NA | NA |
fig <- plot_ly(F_df, x = ~Eje1, y = ~Eje2, z = ~Eje3)
fig <- fig %>% add_markers()
fig <- fig %>% add_text(text = rownames(F_df))
fig <- fig %>% add_markers(x = 0, y = 0, z = 0)
fig <- fig %>% layout(showlegend = FALSE)
fig2.1.8 Individuos suplementarios
Adicionalmente, en los ejes factoriales se pueden representar:
-
Individuos que no participaron en la obtención de los ejes (individuos nuevos)
F_s(i^{+}) = \left\langle \vec{x}_{i^+}, \vec{u}_s \right\rangle = \vec{x}'_{i^{+}} \vec{u}_s
Centro de gravedad de un grupo de individuos, por ejemplo un grupo dado por la categoría de una variable categórica (el valor de una variable cualitativa). Equivalente al centro de gravedad de las coordenadas factoriales de los individuos que pertenecen al grupo.
comer <- as.matrix(cafe[11:12,1:3])
comc <- comer - rep(1,2) %*% t(g) # centrado
comcr <- comc %*% solve(Dsigma) # reducido
colnames(comcr) <- colnames(comer)
Fsup <- comcr %*% U
# primer plano factorial
plot(F[,1:2], las = 1, asp = 1, bty = "n")
text(F[,1:2], label = rownames(F), col = "black", pos = 4)
abline(h = 0, v = 0, col = "darkgrey")
points(Fsup, col="black", pch=20) # cafes comerciales
text(Fsup, labels=c("Com1","Com2"), col="darkgreen", pos = 2)
conta <- factor(c("exce","maiz","ceba","maiz","ceba",
"exce","maiz","ceba","maiz","ceba"))
Fconta <- centroids(F, conta)$centroids
points(Fconta, col = "brown", pch = 20)
text(Fconta, col = "brown", labels = rownames(Fconta), pos = 2)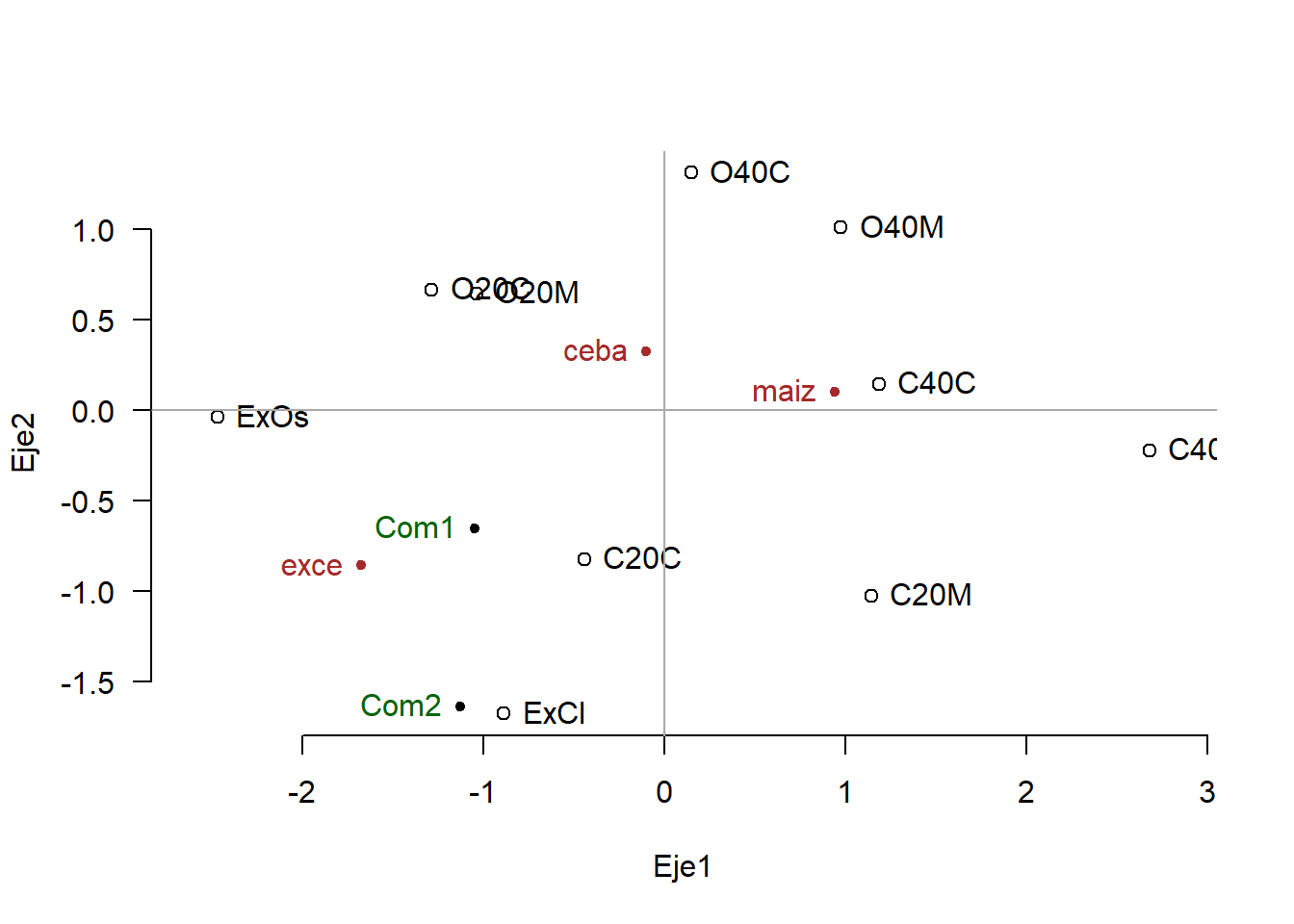
Para los centros de gravedad de las categorías se puede calcular la distancia al origen y los cosenos cuadrados, pero las contribuciones absolutas son cero porque no participan en la obtención de los ejes factoriales.
Adicionalmente, se puede hacer un análisis descriptivo bivariado entre las categorías (variable categórica) y cada uno de los ejes factoriales (variable cuantitativa). Recordemos que la media de un componente principal es cero porque es centrado \left(\mu = 0\right) y su varianza es su valor propio asociado \left(\sigma^2 = \lambda_s\right). Entonces, el valor test para una categoría k, asumida por n_k, estaría dada por, \begin{aligned} \frac{F_s(k) - \mu}{\sigma_k} &= \sqrt{\frac{(n - 1)n_k}{n - n_k}} \frac{F_s(k) - \mu}{\sigma} \\ &= \sqrt{\frac{(n - 1)n_k}{\left(n - n_k\right)\lambda_s}} F_s(k) \end{aligned}
2.2 La nube de variables
Cada punto corresponderá a un vector columna y representará a una variable en un espacio de tantas dimensiones como individuos.
kable(cafe[1:3 ,1:9], digits = 2)| Color | DA | EA | pH | AcidezT | Cafeina | AcidosCl | D2325 | D2272 | |
|---|---|---|---|---|---|---|---|---|---|
| ExCl | 298 | 385.1 | 25 | 5.02 | 11.7 | 1.40 | 2.74 | -0.10 | -0.06 |
| C40M | 361 | 481.3 | 41 | 5.11 | 6.5 | 0.81 | 1.62 | -0.06 | -0.04 |
| C40C | 321 | 422.6 | 40 | 5.12 | 5.8 | 0.80 | 1.61 | -0.06 | -0.04 |
tAux = as.data.frame(t(cafe[1:3 ,1:9]))
fig <- plot_ly(tAux, x = ~ExCl, y = ~C40M, z = ~C40C)
fig <- fig %>% add_markers()
fig <- fig %>% add_text(text = rownames(tAux))
fig <- fig %>% layout(title = "vectores columna\n(variables)",
showlegend = FALSE)
figSea \vec{y}_j, \vec{y_C}_j y \vec{x}_j el j-ésimo vector columna, respectivamente, de las matrices Y, Y_C y X.
Por otra parte, nos será de mucha utilidad el producto interno dado por la matriz \frac{1}{n} I_n, \left\langle \vec{u}, \vec{v} \right\rangle_{\frac{1}{n} I_n} = \vec{u}' \left( \frac{1}{n} I_n \right) \vec{v}
Note que, \begin{aligned} \left\|\vec{1}_n \right\|^2_{\frac{1}{n} I_n} &= \left\langle \vec{1}_n, \vec{1}_n \right\rangle_{\frac{1}{n} I_n} \\ &= \vec{1}_n' \left( \frac{1}{n} I_n \right) \vec{1}_n \\ &= \frac{1}{n} \vec{1}_n' \vec{1}_n \\ &= \frac{1}{n} \sum_{i=1}^n 1^2 \\ &= \frac{1}{n} n \\ &= 1 \end{aligned} es decir, \vec{1}_n es un vector unitario (para el producto interno dado por \frac{1}{n} I_n).
2.2.1 Significado de la media
\begin{aligned} \bar{y}_j &= \frac{1}{n} \sum_{i=1}^n y_{ij} \\ &= \frac{1}{n} \sum_{i=1}^n (y_{ij})(1) \\ &= \frac{1}{n} \vec{y}_j' \vec{1}_n \\ &= \vec{y}_j' \left( \frac{1}{n} I_n \right) \vec{1}_n \\ &= \left\langle \vec{y}_j \, , \, \vec{1}_n \right\rangle_{\frac{1}{n} I_n} \end{aligned} La media de las componentes de la j-ésima variable es la proyección del vector columna sobre la recta que genera el vector unitario \vec{1}_n.
2.2.2 Significado del centrado
Sea \vec{\bar{y}}_j un vector en donde sus n componentes son iguales a \bar{y}_j, entonces \vec{\bar{y}}_j = \bar{y}_j \vec{1}_n y, {\vec{y}_{C}}_j = \vec{y}_j - \vec{\bar{y}}_j Lo que quiere decir que una variable centrada es la proyección de la variable sobre el subespacio ortogonal a la recta generada por \vec{1}_n; subespacio que naturalmente es de una dimensión menos que el de la variable sin centrar.
2.2.3 Significado de las varianzas y covarianzas
\begin{aligned} \sigma_{jk} = cov\left( \vec{y}_j \, , \, \vec{y}_k \right) &= \frac{1}{n}\sum_{i=1}^n \left(y_{ij} - \bar{y}_j \right) \left(y_{ik} - \bar{y}_j\right) \\ &= \left\langle {\vec{y}_{C}}_j \, , \, {\vec{y}_{C}}_k \right\rangle_{\frac{1}{n} I_n} \end{aligned} de donde, \begin{aligned} \sigma^2_{j} = var\left( \vec{y}_j \right) &= \frac{1}{n}\sum_{i=1}^n \left( y_{ij} - \bar{y}_j \right)^2 \\ &= \left\langle {\vec{y}_{C}}_j \, , \, {\vec{y}_{C}}_j \right\rangle_{\frac{1}{n} I_n} \\ &= \left\| {\vec{y}_{C}}_j \right\|^2_{\frac{1}{n} I_n} \end{aligned} La covarianza es el producto interno entre vectores columna y la varianza es la norma al cuadrado del vector columna (la desviación estándar es la norma).
2.2.4 Significado de la reducción
\begin{aligned} \vec{x}_j &= \frac{\vec{y}_j - \vec{\bar{y}}_j}{\sigma_j} \\ &= \frac{{\vec{y}_{C}}_j}{\left\| {\vec{y}_{C}}_j \right\|} \end{aligned} Un vector columna después del centrado y reducido es un vector unitario \left\| \vec{x}_j \right\| = 1.
2.2.5 Significado de la correlación
\begin{aligned} \rho_{jk} = cor\left( \vec{y}_j \, , \, \vec{y}_k \right) &= \frac{\sigma_{jk}}{\sigma_{j} \sigma_{k}} \\ &= \frac{\left\langle {\vec{y}_{C}}_j \, , \, {\vec{y}_{C}}_k \right\rangle_{\frac{1}{n} I_n}}{\left\| {\vec{y}_{C}}_j \right\|_{\frac{1}{n} I_n} \left\| {\vec{y}_{C}}_k \right\|_{\frac{1}{n} I_n}} \\ &= \cos\left(\angle_{{\vec{y}_{C}}_j}^{{\vec{y}_{C}}_k}\right) \\ &= \cos\left(\angle_{\vec{x}_j}^{\vec{x}_k}\right) \end{aligned}
Concluimos que la nube de variables, luego del centrado y el reducido, es una representación de la matriz de correlaciones. Por ejemplo, vectores columna ortogonales/perpendiculares indican que las respectivas variables no están correlacionadas.
2.2.6 Inercia
\begin{aligned} Inercia(N_p) &= \sum_{j=1}^{p} m_j \, d^2_{\frac{1}{n} I_n}\left(\vec{y}_{Ci} ,\vec{0}\right) \end{aligned}
Cuando todos los m_j son iguales a 1, \begin{aligned} Inercia(N_p) &= \sum_{j=1}^{p} m_j \, d^2_{\frac{1}{n} I_n}\left(\vec{y}_{Ci} ,\vec{0}\right) \\&= \sum_{j=1}^{p} \left\| {\vec{y}_{C}}_j \right\|^2_{\frac{1}{n} I_n} \\ &= \sum_{j=1}^{p}var\left( \vec{y}_j \right) \\ &= \sum_{j=1}^{p} \sigma_j^2 \end{aligned}
La contribución del vector columna a la inercia de la nube de variables es la varianza de la variable correspondiente. En el caso normado (centrado y reducido) cada variable aporta 1 a la inercia y la inercia total es igual al número de variables.
2.2.7 Nuevos ejes
La proyección de una variable \vec{x}_j sobre una recta generada por el vector v (\frac{1}{n} I_n unitario) en \mathbb{R}^n es, \begin{aligned} \left\langle \vec{x}_j \, , \, \vec{v} \right\rangle_{\frac{1}{n} I_n} &= \vec{x}_j' \left( \frac{1}{n} I_n \right) \vec{v} \\ &= \frac{1}{n} \vec{x}_j' \vec{v} \end{aligned}
La inercia de las p variables proyectadas es, \begin{aligned} \sum_{j=1}^p \left( \frac{1}{n} \vec{x}_j' \vec{v} \right)^2 &= \frac{1}{n^2} \left( X \vec{v} \right)'\left( X \vec{v} \right) \\ &= \frac{1}{n^2} \vec{v}' X X' \vec{v} \\ &= \frac{1}{n} \left[ \vec{v}' \left( \frac{1}{n} X X' \right) \vec{v} \right] \end{aligned}
El eje de mayor inercia proyectada se encuentra solucionando, \begin{aligned} {\underset {v \in \mathbb{R}^n}{\text{arg max}}}\quad & \frac{1}{n} \left[ \vec{v}' \left( \frac{1}{n} X X' \right) \vec{v} \right] \\ {\text{subject to}}\quad &\vec{v}' \left( \frac{1}{n} I_n \right) \vec{v} - 1 = 0. \end{aligned}
Usando multiplicadores de Lagrange, f(u) = \frac{1}{n} \left[ \vec{v}' \left( \frac{1}{n} X X' \right) \vec{v} \right] - \mu \left( \frac{1}{n} \vec{v}' \vec{v} - 1 \right) que tiene como primera derivada, \frac{d}{du} f(u) = \frac{2}{n} \left[ \left( \frac{1}{n} X X' \right) v \right] - \frac{2}{n} \mu v Igualando a cero, \begin{aligned} \frac{2}{n} \left[ \left( \frac{1}{n} X X' \right) v \right] - \frac{2}{n} \mu v &= 0 \\ \left( \frac{1}{n} X X' \right) v &= \mu v \end{aligned} De donde concluimos que la solución está dada por el vector propio (o su inverso aditivo) asociado al mayor valor propio de la matriz \frac{1}{n} X X'. Sin embargo, los valores propios mayores que cero de \frac{1}{n} X' X y \frac{1}{n} X X' son iguales.
La coordenada sobre un eje s de la variable j, se obtiene mediante, G_s(j) = \frac{1}{n} \vec{x}_j' v_s
2.2.8 Relación entre las dos nubes (espacios)
La matriz \frac{1}{n} XX' tiene p valores propios, que son iguales a los valores propios de \frac{1}{n} X'X y los restantes n - p valores propios son 0.
El vector coordenadas de los n individuos sobre el eje s, F_s, es un vector propio de \frac{1}{n} XX'.
La varianza de F_s es \lambda_s, y por lo tanto, el vector propio v_s se puede calcular mediante: v_s = \frac{1}{\sqrt{\lambda_s}} F_s.
G_s, vector de coordenadas de las p variables sobre el eje s, es un vector propio de \frac{1}{n} X'X.
La varianza de G_s es \lambda_s, y por lo tanto, se puede obtener mediante: G_s = \sqrt{\lambda_s} u_s.
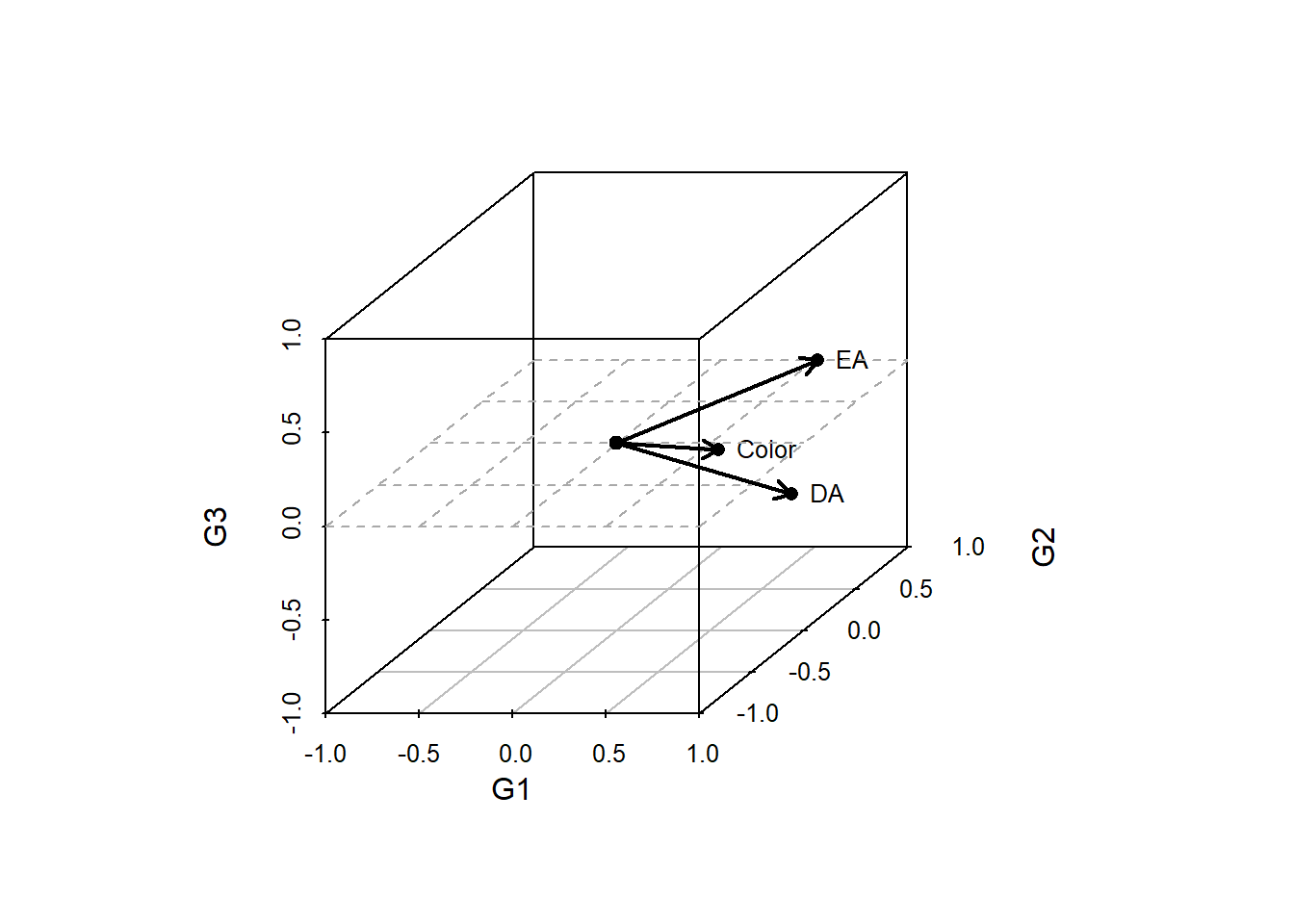
En el ACP normado, las coordenadas de G_s son las correlaciones entre las variables y el eje s.
| G1 | G2 | G3 | |
|---|---|---|---|
| Color | 0.833 | -0.518 | 0.194 |
| DA | 0.967 | -0.061 | -0.246 |
| EA | 0.661 | 0.741 | 0.115 |
kable(as.data.frame(cor(Y,F)), digits = 3)| Eje1 | Eje2 | Eje3 | |
|---|---|---|---|
| Color | 0.833 | -0.518 | 0.194 |
| DA | 0.967 | -0.061 | -0.246 |
| EA | 0.661 | 0.741 | 0.115 |
G3D <- scatterplot3d(G, pch = 16, color = "black",
xlim = c(-1,1), ylim = c(-1,1),
zlim = c(-1,1), asp = 1)
coord <- G3D$xyz.convert(G)
text(coord, labels = rownames(G),
cex = 0.8, col = "black", pos = 4)
G3D$plane(0, 0, 0, col="darkgrey")
G3D$points3d(t(c(0,0,0)), pch = 19, col = "black")
cero <- G3D$xyz.convert(0,0,0)
for(eje in 1:3){
arrows(cero$x, cero$y, coord$x[eje], coord$y[eje],
lwd = 2, length = 0.1, col = "black")
}
# Código experimental
G_df = as.data.frame(G)
fig <- plot_ly(G_df, x = ~G1, y = ~G3, z = ~G2)
# Graficare como puntos pero sería mejor como flechas
fig <- fig %>% add_markers()
fig <- fig %>% add_text(text = rownames(G_df))
fig <- fig %>% add_markers(x = 0, y = 0, z = 0)
sphere <- function(n = 100){
theta <- seq(0, pi, length.out = n)
phi <- seq(0, 2 * pi, length.out = n)
r <- 1
x <- r * outer(sin(theta), cos(phi))
y <- r * outer(sin(theta), sin(phi))
z <- r * outer(cos(theta), rep(1, length(phi)))
return(list(x = x, y = y, z = z))
}
s_c <- sphere()
fig <- fig %>% add_trace(x = s_c$x, y = s_c$y, z = s_c$z,
type = 'surface', opacity = 0.2,
colorscale = "Greys", # cambiar por valor fijo
showscale = FALSE)
fig <- fig %>% layout(title = "vectores columna\n(variables)",
scene = list(xaxis = list(range = c(-1,1)),
yaxis = list(range = c(-1,1)),
zaxis = list(range = c(-1,1)),
aspectratio = list(x=1,y=1,z=1),
camera = list(eye = list(x=1,y=1,z=1))),
showlegend = FALSE)
fig2.2.9 Ayudas interpretación
De manera equivalente a como se hizo para los individuos, para las variables también se pueden calcular contribuciones absolutas y cosenos cuadrados. En el caso del ACP normado, la distancia al origen de cada una de las variables siempre es igual a 1.
# Primer plano factorial
s.corcircle(G)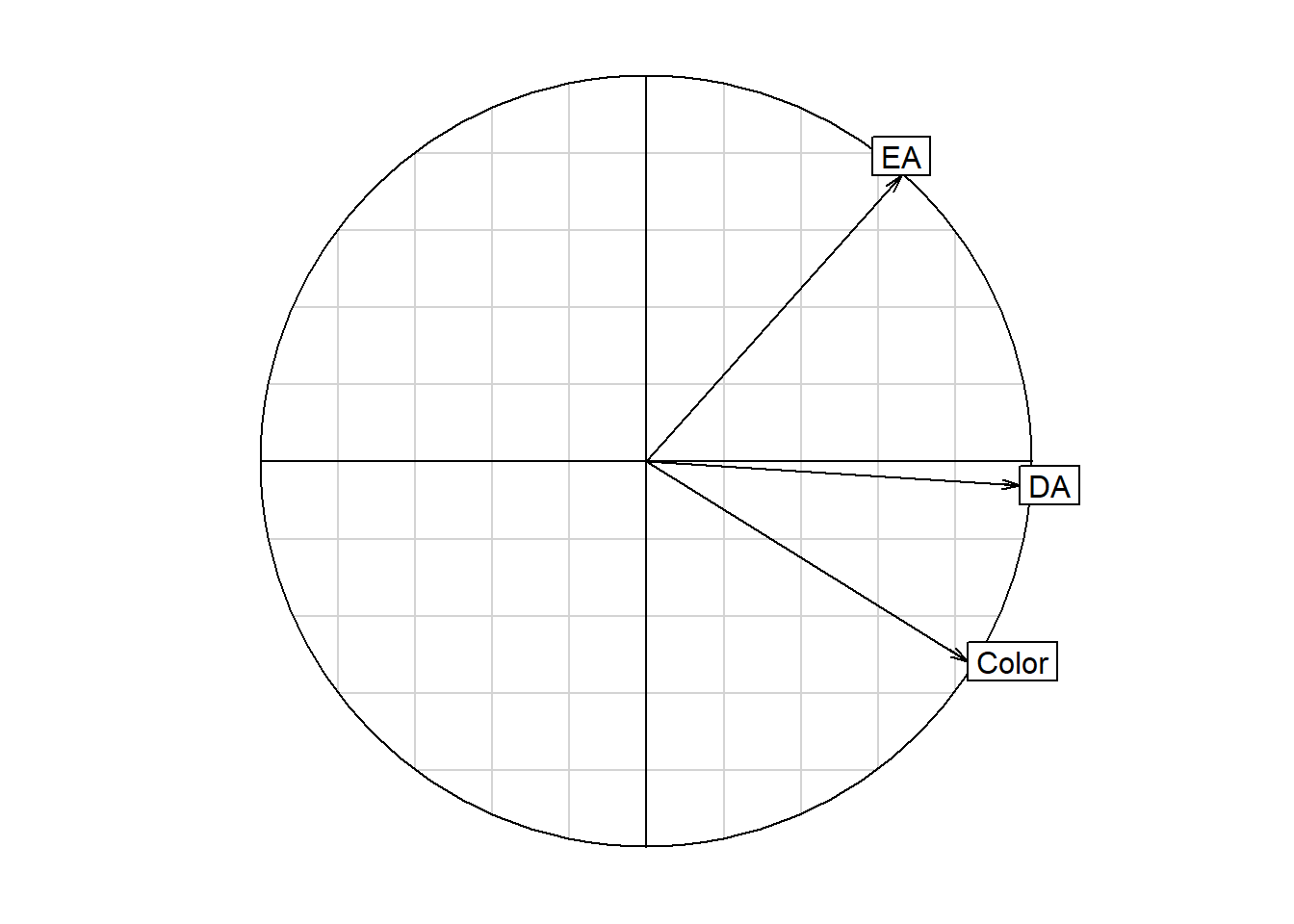
kable(as.data.frame(V), digits = 2)| Color | DA | EA | |
|---|---|---|---|
| Color | 1.00 | 0.79 | 0.19 |
| DA | 0.79 | 1.00 | 0.57 |
| EA | 0.19 | 0.57 | 1.00 |
2.2.10 Variables continuas suplementarias
Sobre los ejes principales se pueden proyectar variables que no participaron en el análisis. Igual que como ocurre para las demás variables, cada coordenada es la correlación entre la variable y cada eje.
En el ejemplo “Café” se proyecta la nota de impresión global dada por un panel de catadores para explorar su relación con las tres variables físicas en conjunto.
s.corcircle(G)
# proyeccion de nota como variable ilustrativa
Impresion <- cafe[1:10,16]
FImpresion <- cor(Impresion, F)
arrows(0, 0, FImpresion[1], FImpresion[2],
col = "black", angle = 10, lty = 3)
text(FImpresion, "Impresion", col="black",
pos = 1, font = 3)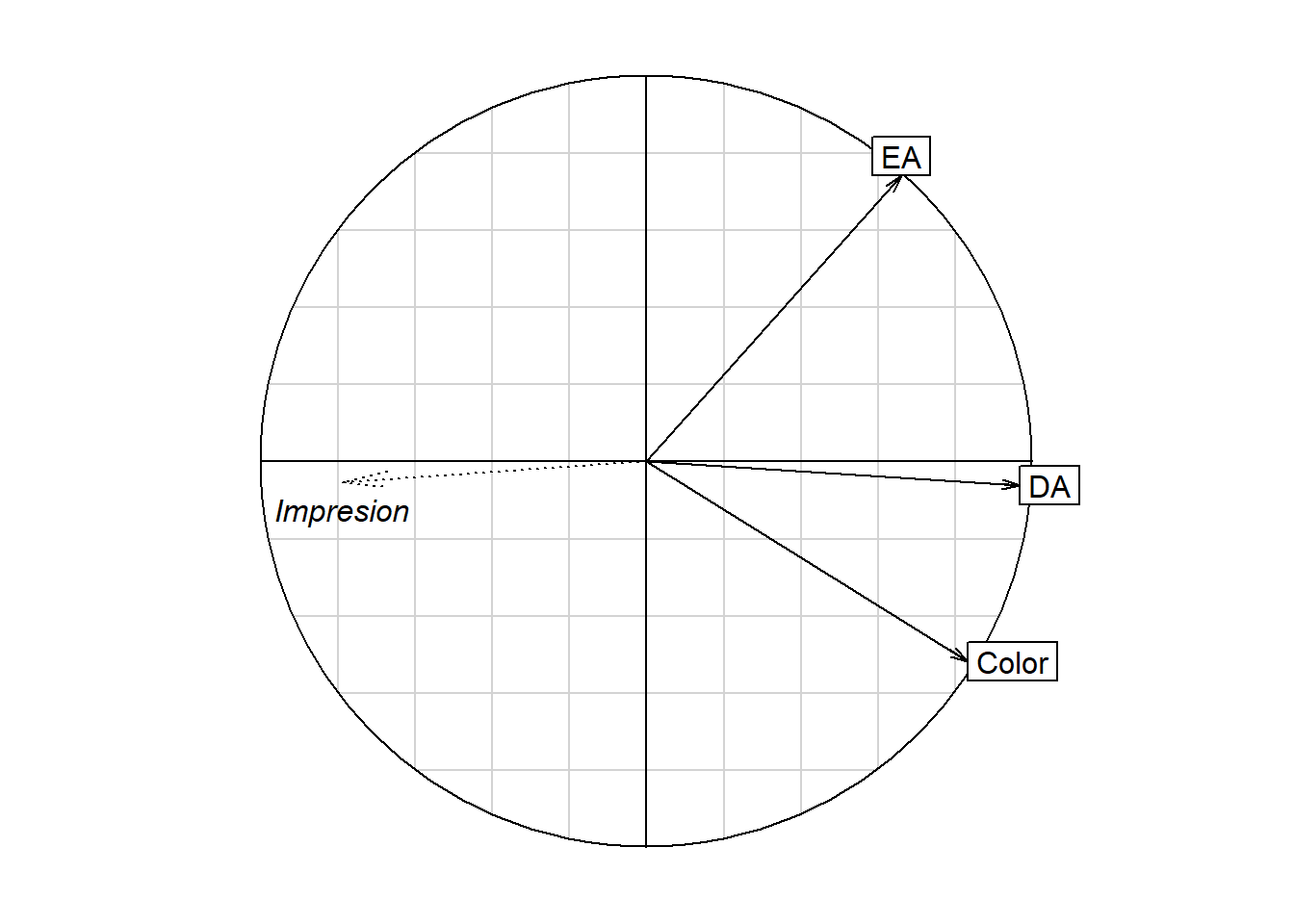
2.3 ACP con ade4 y FactoClass
| Función | Librería | Descripción |
|---|---|---|
dudi.pca |
ade4 |
ACP, por defecto normado. |
inertia.dudi |
ade4 |
Ayudas para la interpretación, por defecto solo valores propios. |
s.corcircle |
ade4 |
Círculos de correlaciones. |
plotcc |
FactoClass |
Círculos de correlaciones con ggplot2. |
s.arrow |
ade4 |
Planos factoriales de las variables, en un ACP no normado. |
s.label |
ade4 |
Planos factoriales de los individuos. |
plot.dudi |
FactoClass |
Planos factoriales de los individuos, usando el parámetro Tcol = FALSE. |
suprow |
ade4 |
Coordenadas de individuos suplementarios. |
points |
graphics |
Proyectar puntos en un gráfico. |
text |
graphics |
Poner etiquetas asociadas a los puntos en un gráfico. |
cor(acp$li,Xsup) |
stats |
Correlaciones entre variables suplementarias y ejes (coordenadas en un ACP normado. Si ACP es el objeto de salida del ACP y Xsup es la tabla de variables suplementarias. |
arrows |
graphics |
Proyectar variables ilustrativas sobre un círculo de correlaciones. |
supqual |
FactoClass |
Coordenadas y ayudas a la interpretación de variables cualitativas ilustrativas. |
s.class |
ade4 |
Planos factoriales de los individuos destacando las clases de una partición definida por una variable cualitativa. |
# ACP normado con variables físicas y reteniendo dos ejes
acp <- dudi.pca(cafe[1:10,1:3], scannf=FALSE)
acp$cent # medias de las variables:Color DA EA
276.7 401.2 35.5 round(acp$norm, 2) # desviación estándar de las variablesColor DA EA
55.73 39.50 5.78 inertia(acp) # valores propios y porcentajesInertia information:
Call: inertia.dudi(x = acp)
Decomposition of total inertia:
inertia cum cum(%)
Ax1 2.0670 2.067 68.90
Ax2 0.8216 2.889 96.29
Ax3 0.1113 3.000 100.00barplot(acp$eig) # histograma de valores propios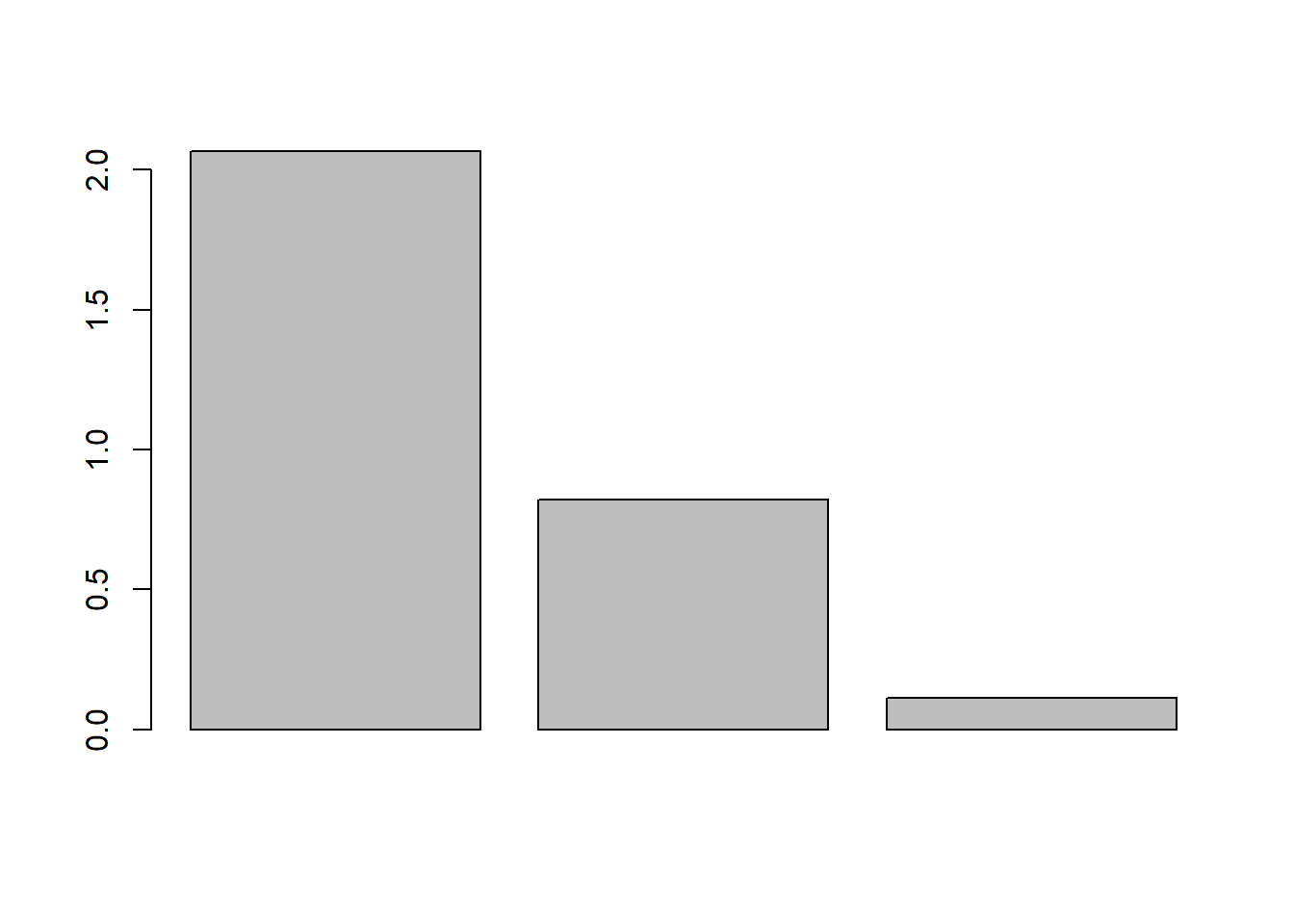
round(acp$c1, 3) # vectores propios CS1 CS2
Color 0.579 -0.571
DA 0.673 -0.067
EA 0.460 0.818Código
fviz_screeplot(acp, addlabels = TRUE,
ylim = c(0, 100))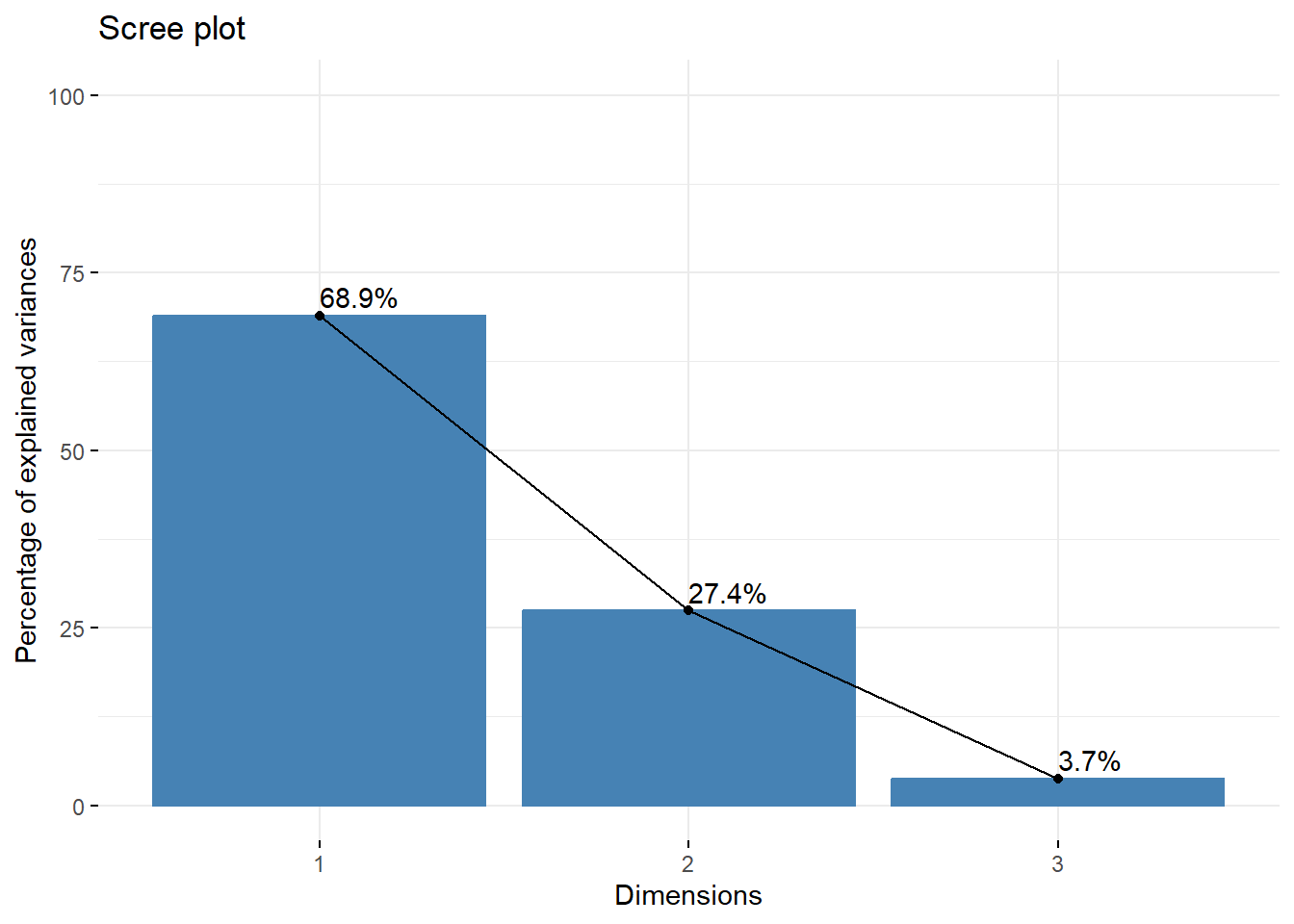
s.corcircle(acp$co)
# proyeccion de la variable Impresion como ilustrativa
(coimpre <- cor(cafe[1:10,16], acp$li)) Axis1 Axis2
[1,] -0.7887 -0.05268s.arrow(coimpre, label="Impresion",
add.plot = TRUE, boxes = FALSE)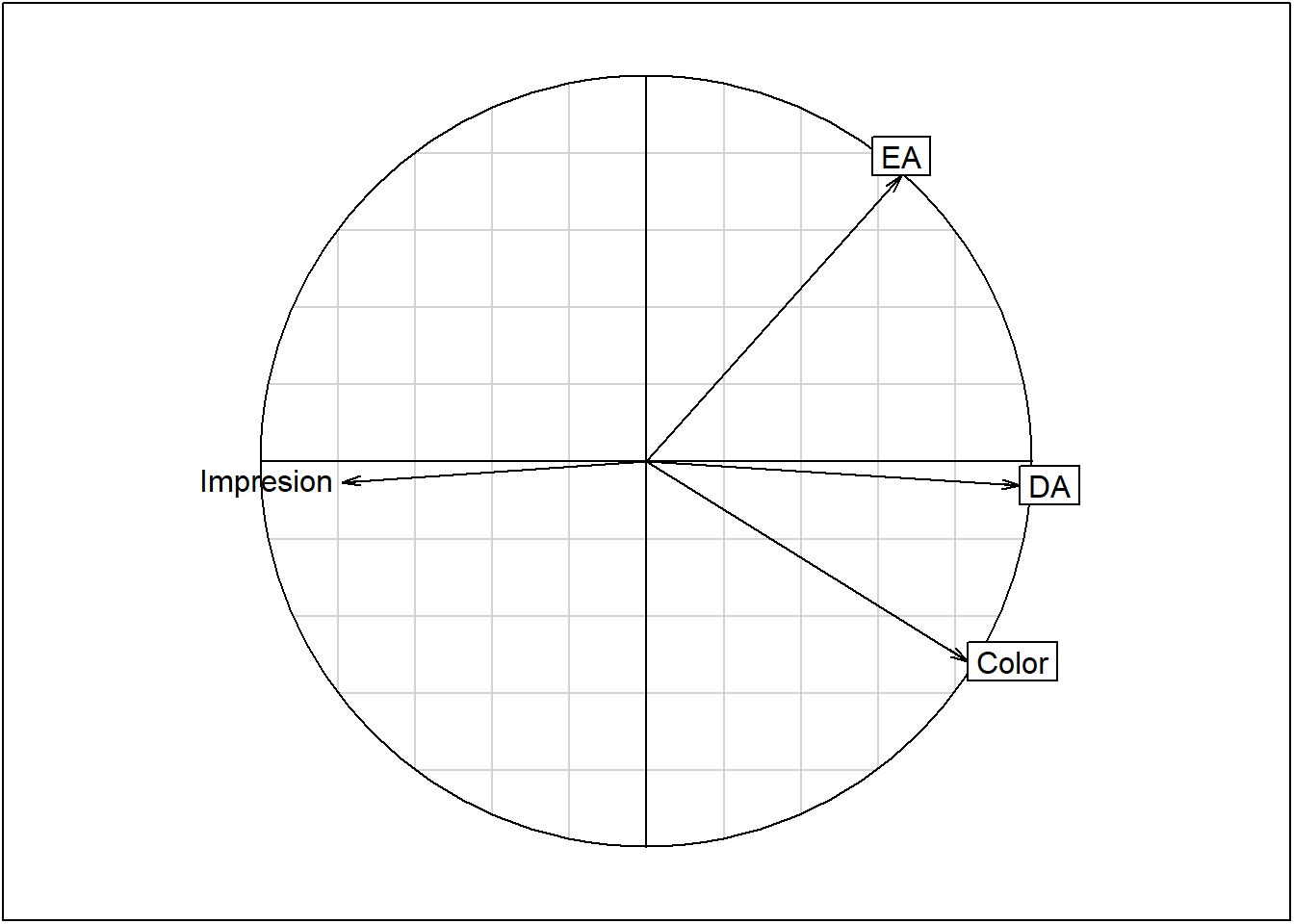
# coordenadas de las variables = correlaciones con los ejes
round(acp$co, 3) Comp1 Comp2
Color 0.833 -0.518
DA 0.967 -0.061
EA 0.661 0.741# ayudas para la interpretación de las variables
inertia(acp, , TRUE)Inertia information:
Call: inertia.dudi(x = acp, col.inertia = TRUE)
Decomposition of total inertia:
inertia cum cum(%)
Ax1 2.0670 2.067 68.90
Ax2 0.8216 2.889 96.29
Ax3 0.1113 3.000 100.00
Column contributions (%):
Color DA EA
33.33 33.33 33.33
Column absolute contributions (%):
Axis1 Axis2
Color 33.58 32.6507
DA 45.28 0.4463
EA 21.14 66.9029
Signed column relative contributions:
Axis1 Axis2
Color 69.41 -26.8274
DA 93.59 -0.3667
EA 43.70 54.9706
Cumulative sum of column relative contributions (%):
Axis1 Axis1:2 Axis3:3
Color 69.41 96.24 3.759
DA 93.59 93.96 6.042
EA 43.70 98.67 1.331fig <- fviz_pca_var(acp, col.var = "black")
coord <- cor(cafe[1:10,16], F)
rownames(coord) <- "Impresión"
fviz_add(fig, coord, color ="blue", geom="arrow", labelsize=3)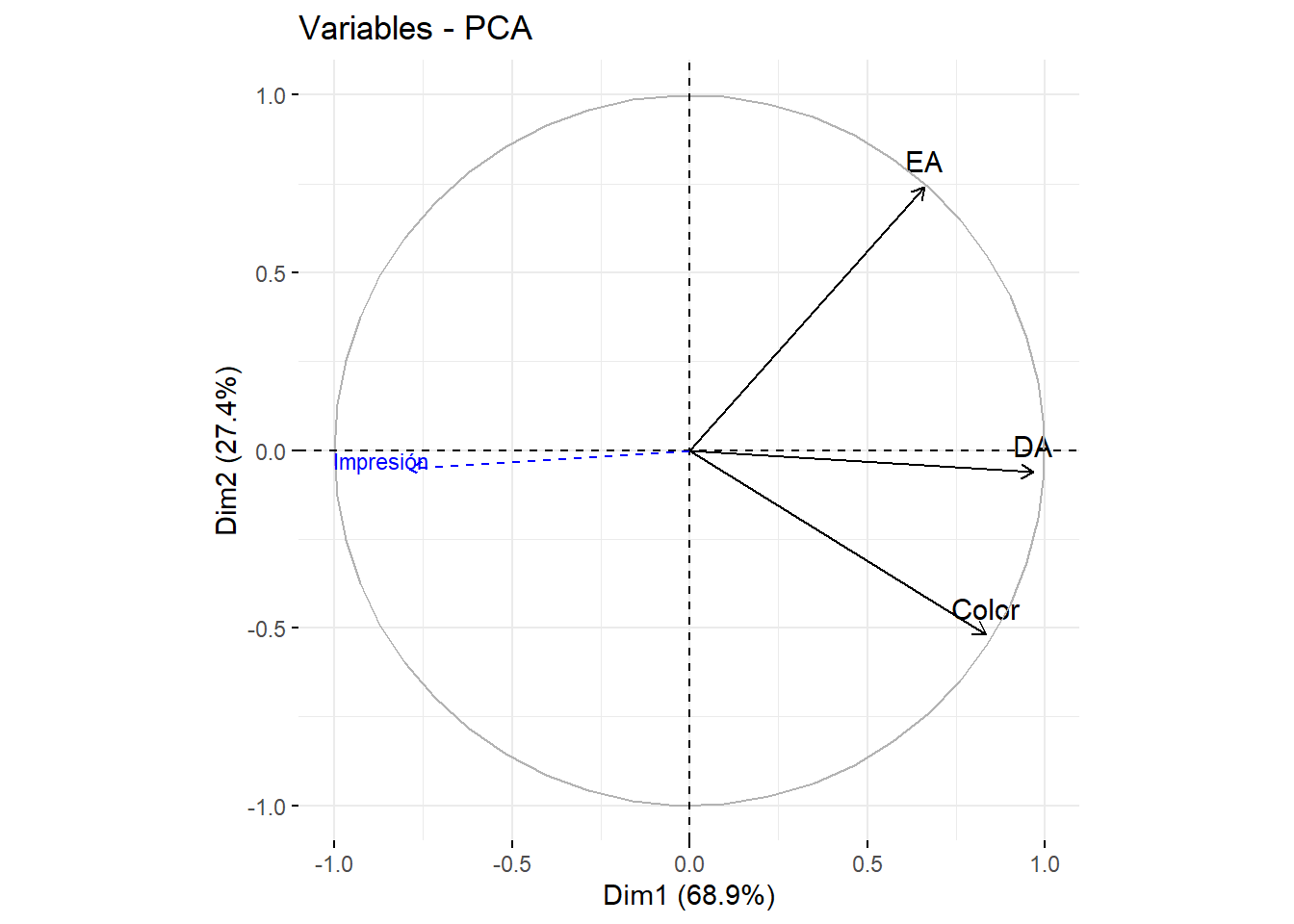
plot(acp, Tcol = FALSE, gg = TRUE)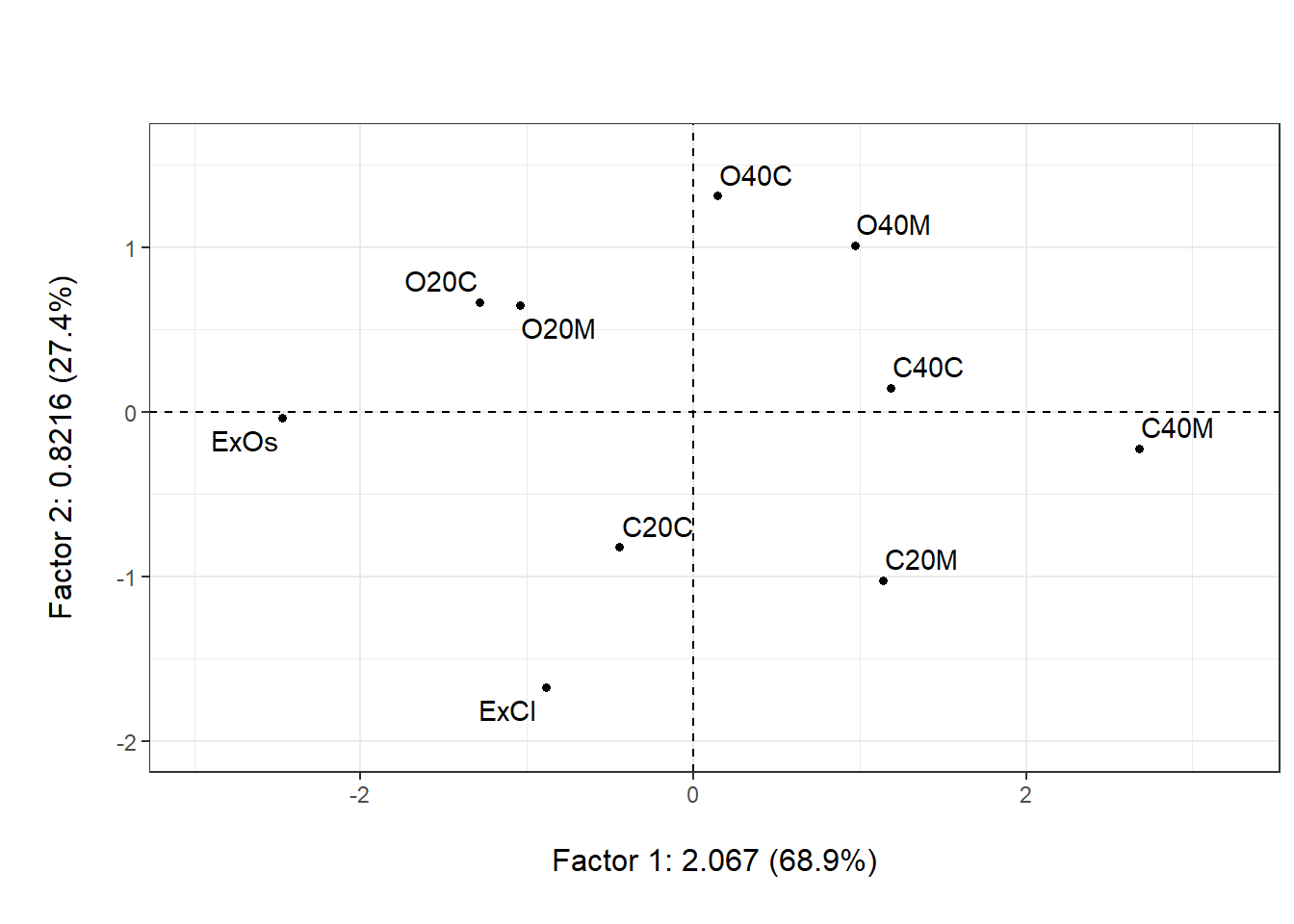
round(acp$li, 2) # coordenadas de los cafés Axis1 Axis2
ExCl -0.89 -1.68
C40M 2.68 -0.22
C40C 1.18 0.15
C20M 1.14 -1.02
C20C -0.44 -0.82
ExOs -2.47 -0.04
O40M 0.98 1.01
O40C 0.15 1.31
O20M -1.04 0.65
O20C -1.29 0.67inertia(acp, TRUE) # ayudas para la interpretación de los cafésInertia information:
Call: inertia.dudi(x = acp, row.inertia = TRUE)
Decomposition of total inertia:
inertia cum cum(%)
Ax1 2.0670 2.067 68.90
Ax2 0.8216 2.889 96.29
Ax3 0.1113 3.000 100.00
Row contributions (%):
ExCl C40M C40C C20M C20C ExOs O40M O40C O20M O20C
12.025 24.363 5.106 8.247 4.965 20.794 6.589 5.825 5.035 7.051
Row absolute contributions (%):
Axis1 Axis2
ExCl 3.8050 34.19476
C40M 34.7227 0.60012
C40C 6.7777 0.25918
C20M 6.3136 12.76906
C20C 0.9510 8.23410
ExOs 29.4872 0.01798
O40M 4.5990 12.44216
O40C 0.1027 20.98013
O20M 5.2368 5.07039
O20C 8.0044 5.43211
Signed row relative contributions:
Axis1 Axis2
ExCl -21.803 -77.88530
C40M 98.199 -0.67464
C40C 91.455 1.39017
C20M 52.749 -42.40673
C20C -13.197 -45.42087
ExOs -97.704 -0.02368
O40M 48.090 51.71557
O40C 1.214 98.64686
O20M -71.665 27.58164
O20C -78.219 21.10044
Cumulative sum of row relative contributions (%):
Axis1 Axis1:2 Axis3:3
ExCl 21.803 99.69 0.3122
C40M 98.199 98.87 1.1262
C40C 91.455 92.84 7.1552
C20M 52.749 95.16 4.8442
C20C 13.197 58.62 41.3821
ExOs 97.704 97.73 2.2724
O40M 48.090 99.81 0.1949
O40C 1.214 99.86 0.1387
O20M 71.665 99.25 0.7532
O20C 78.219 99.32 0.6803lcom <- suprow(acp, cafe[11:12,1:3])$lisup
plot(acp, Tcol = FALSE)
points(lcom, col = "darkgreen")
text(lcom, rownames(lcom), col = "darkgreen", pos = 2)
# proyección variables conta (contaminación) como ilustrativa
conta<-factor(c("exce","maiz","ceba","maiz","ceba",
"exce","maiz","ceba","maiz","ceba"))
supconta <- supqual(acp, conta)
points(supconta$coor, col = "red", pch = 20)
text(supconta$coor, rownames(supconta$coor),
col = "red", pos = 2)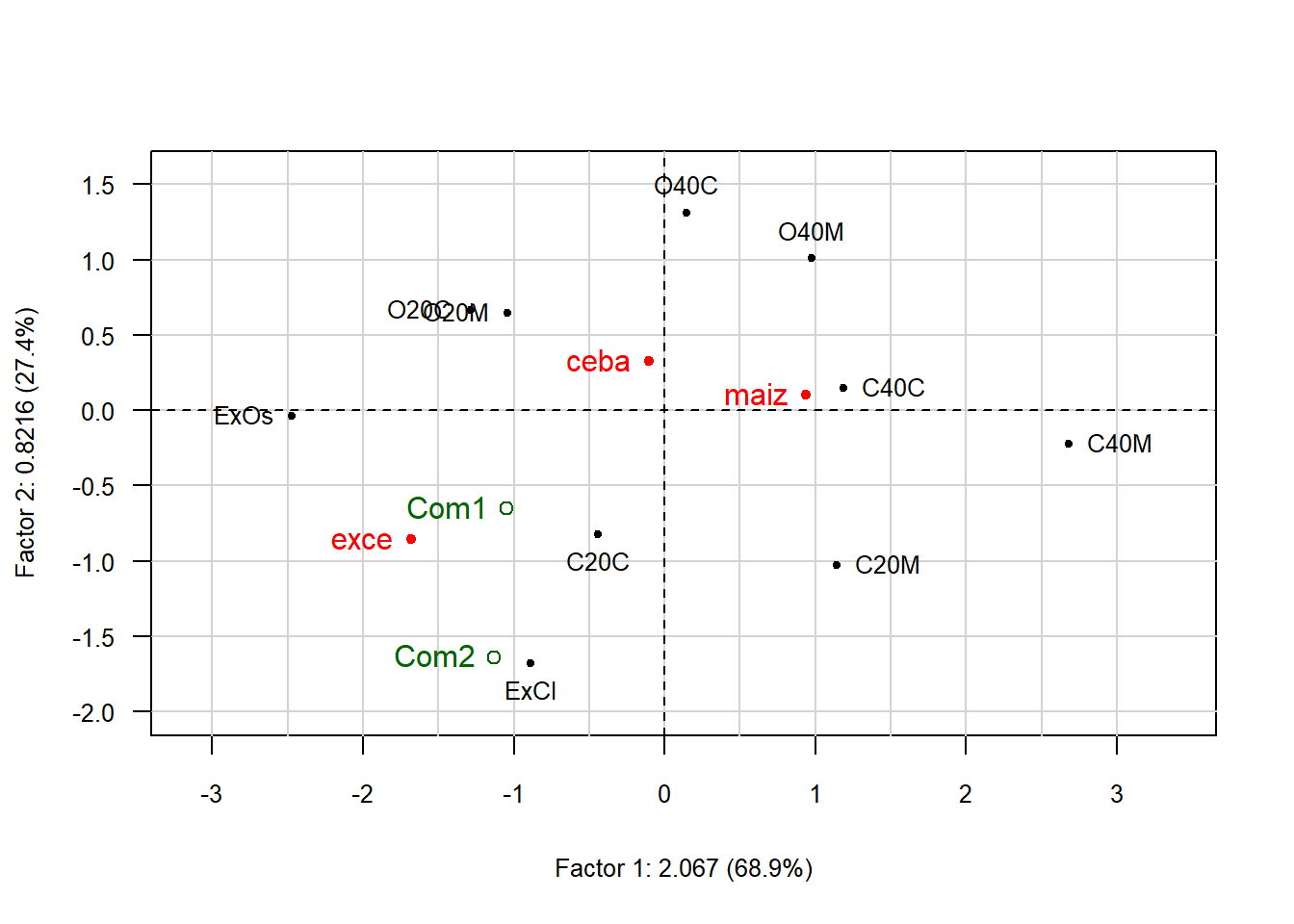
round(supconta$tv, 2); # valores test de categorías de conta Axis1 Axis2
ceba -0.17 0.88
exce -1.75 -1.42
maiz 1.60 0.28plot(acp, Tcol = FALSE)
ade4::s.class(acp$li, conta, col = c("orange","darkgreen","red"),
add.plot = TRUE, cellipse = 0, clabel = 0.5)
lcom <- suprow(acp, cafe[11:12,1:3])$lisup
points(lcom, col = "blue", pch = 16, cex = .8)
text(lcom, rownames(lcom), col = "blue", pos = 2, cex = .8)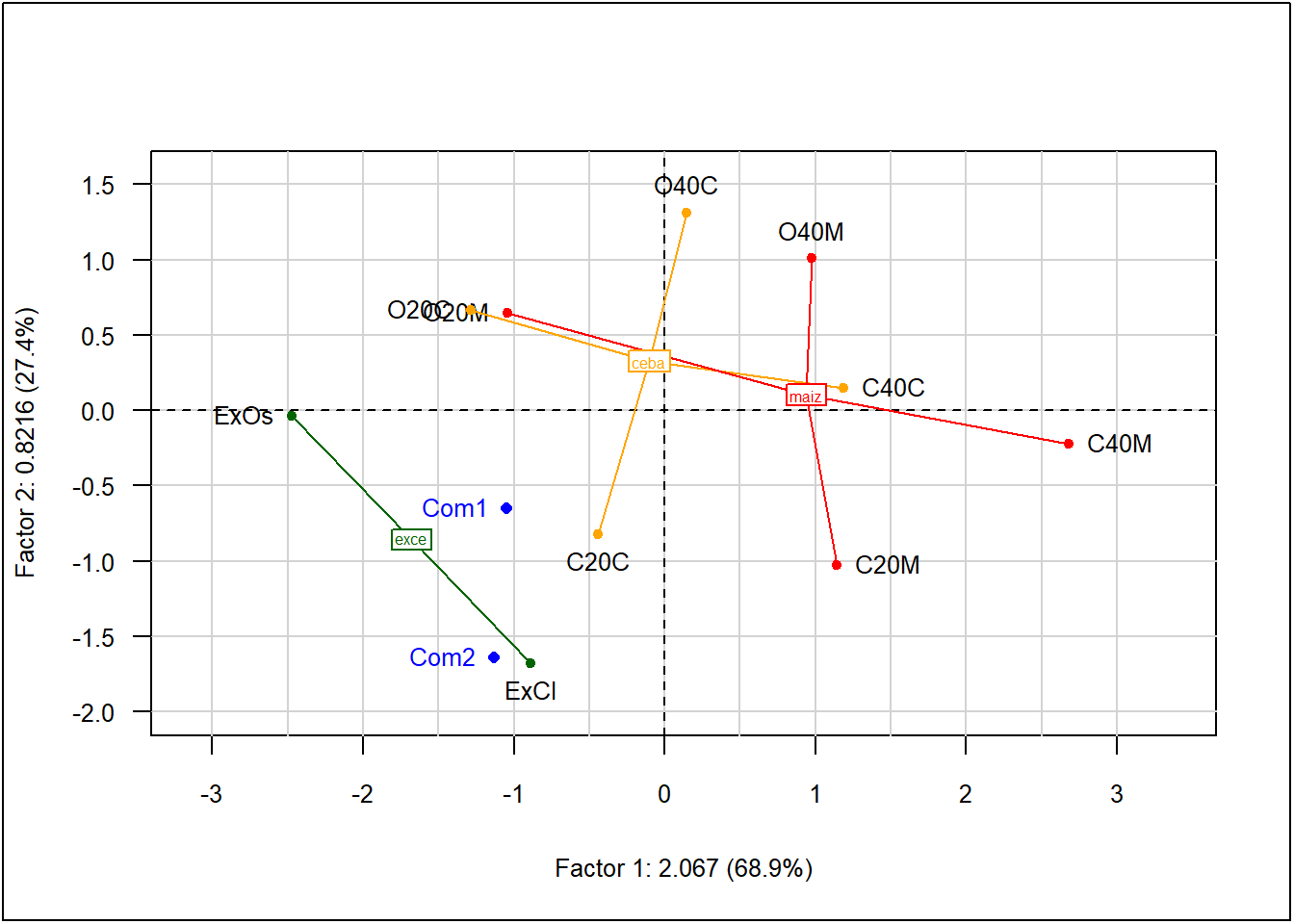
Podemos unir en un solo gráfico:
fig <- fviz_pca_biplot(acp, repel = TRUE)
fig <- fviz_add(fig, coord, color ="blue", geom="arrow")
fviz_add(fig, lcom, color ="purple", geom="point")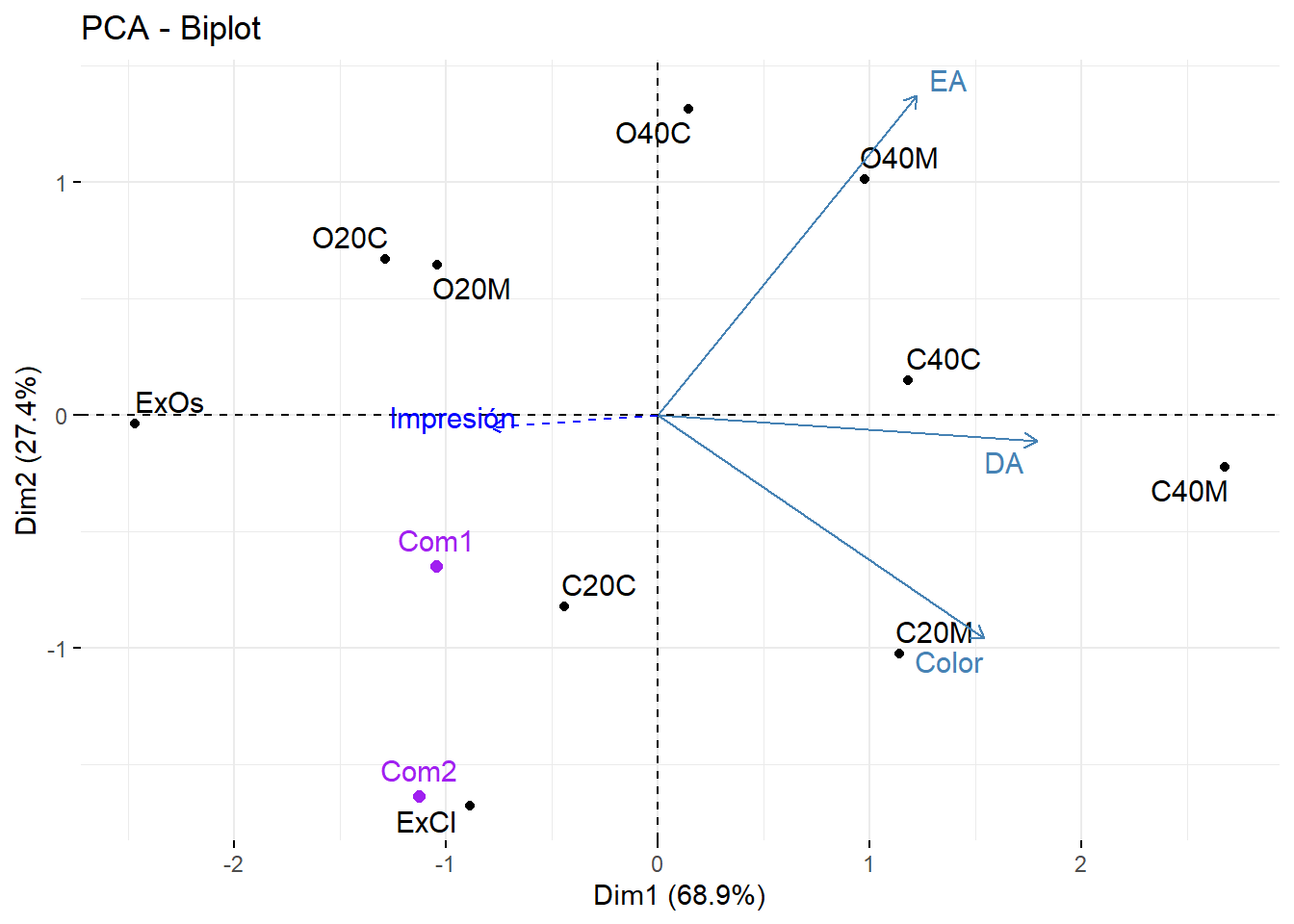
¿Cuál sería el resultado si hubiésemos usado más variables (por ejemplo, todas las físico-químicas)?
Código
library(Factoshiny) # cargar Factoshiny
res <- PCAshiny(cafe) # activar ACP para cafe2.4 Ejemplo (ACP de examen de admisión)
Ejemplo de aplicación de ACP: resultados del examen de admisión a las carreras de la Facultad de Ciencias
carr mate cien soci text
Biol:63 Min. : 9.3 Min. : 9.37 Min. : 9.02 Min. : 8.2
Esta:66 1st Qu.:10.9 1st Qu.:10.84 1st Qu.:10.70 1st Qu.:10.9
Farm:73 Median :11.5 Median :11.48 Median :11.36 Median :11.2
Fisi:82 Mean :11.8 Mean :11.59 Mean :11.36 Mean :11.4
Geol:45 3rd Qu.:12.3 3rd Qu.:12.12 3rd Qu.:11.71 3rd Qu.:11.9
Mate:53 Max. :18.3 Max. :16.52 Max. :14.84 Max. :16.5
Quim:63
imag exam gene estr orig edad
Min. : 8.48 Min. : 477 F:128 bajo :179 Bogo:311 a16m:118
1st Qu.:10.68 1st Qu.: 667 M:317 medio:185 Cund: 38 a17 :171
Median :11.26 Median : 710 alto : 81 Otro: 96 a18 : 56
Mean :11.30 Mean : 718 a19M:100
3rd Qu.:12.01 3rd Qu.: 761
Max. :14.71 Max. :1151
niLE niMa stra age
siLE: 46 siMa:315 E0: 2 Min. :15.0
noLE:399 noMa:130 E1: 36 1st Qu.:16.0
E2:141 Median :17.0
E3:185 Mean :18.1
E4: 72 3rd Qu.:18.0
E5: 8 Max. :44.0
E6: 1 Y <- admi[, 2:6]
plotpairs(Y)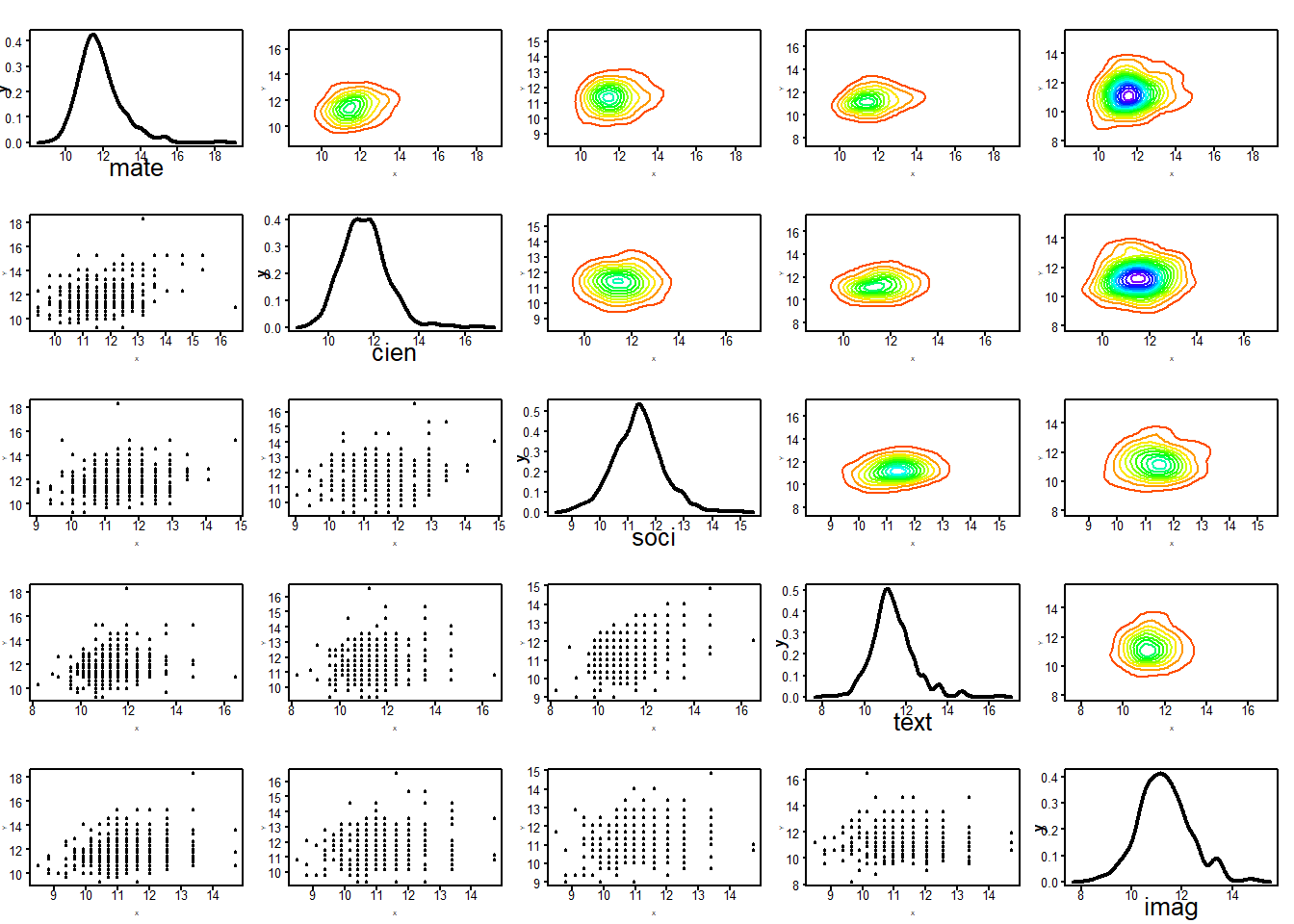
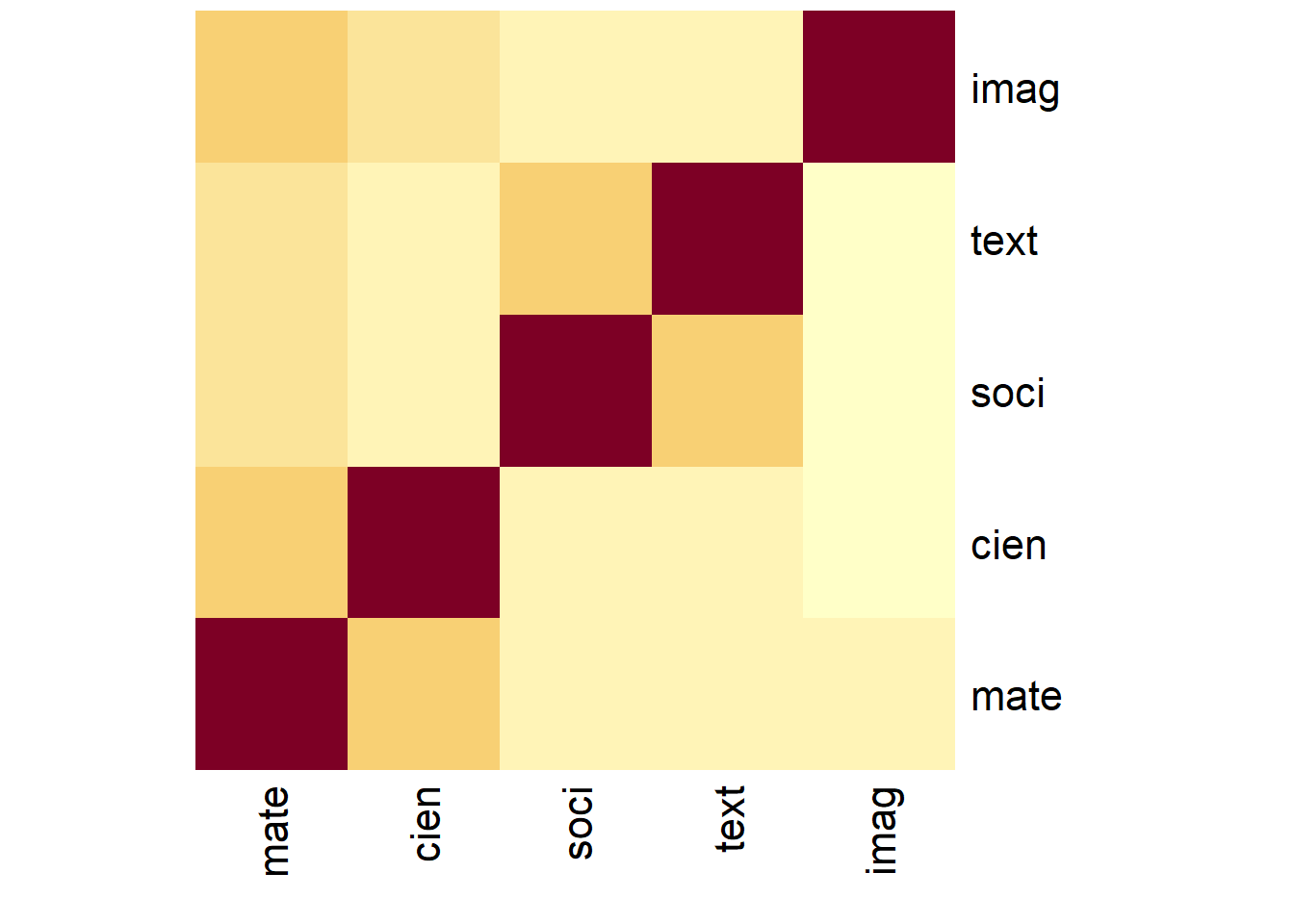
| mate | cien | soci | text | imag | |
|---|---|---|---|---|---|
| mate | 1.000 | 0.341 | 0.242 | 0.242 | 0.211 |
| cien | 0.341 | 1.000 | 0.160 | 0.202 | 0.123 |
| soci | 0.242 | 0.160 | 1.000 | 0.372 | 0.105 |
| text | 0.242 | 0.202 | 0.372 | 1.000 | 0.046 |
| imag | 0.211 | 0.123 | 0.105 | 0.046 | 1.000 |
ACP:
Código
# Con Factoshiny (y FactoMineR):
library(Factoshiny) # cargar Factoshiny
res <- PCAshiny(admi) # activar ACP para admi# Con ade4 y FactoClass
acp <- dudi.pca(Y, scannf = FALSE, nf = 3)
valp <- t(inertia(acp)$tot.inertia) # valores propios
kable(valp, digits = 3)| Ax1 | Ax2 | Ax3 | Ax4 | Ax5 | |
|---|---|---|---|---|---|
| inertia | 1.852 | 1.025 | 0.870 | 0.638 | 0.616 |
| cum | 1.852 | 2.877 | 3.746 | 4.384 | 5.000 |
| cum(%) | 37.034 | 57.532 | 74.925 | 87.686 | 100.000 |
Código
# Con factoextra:
fviz_screeplot(acp, addlabels = TRUE, ylim = c(0, 50))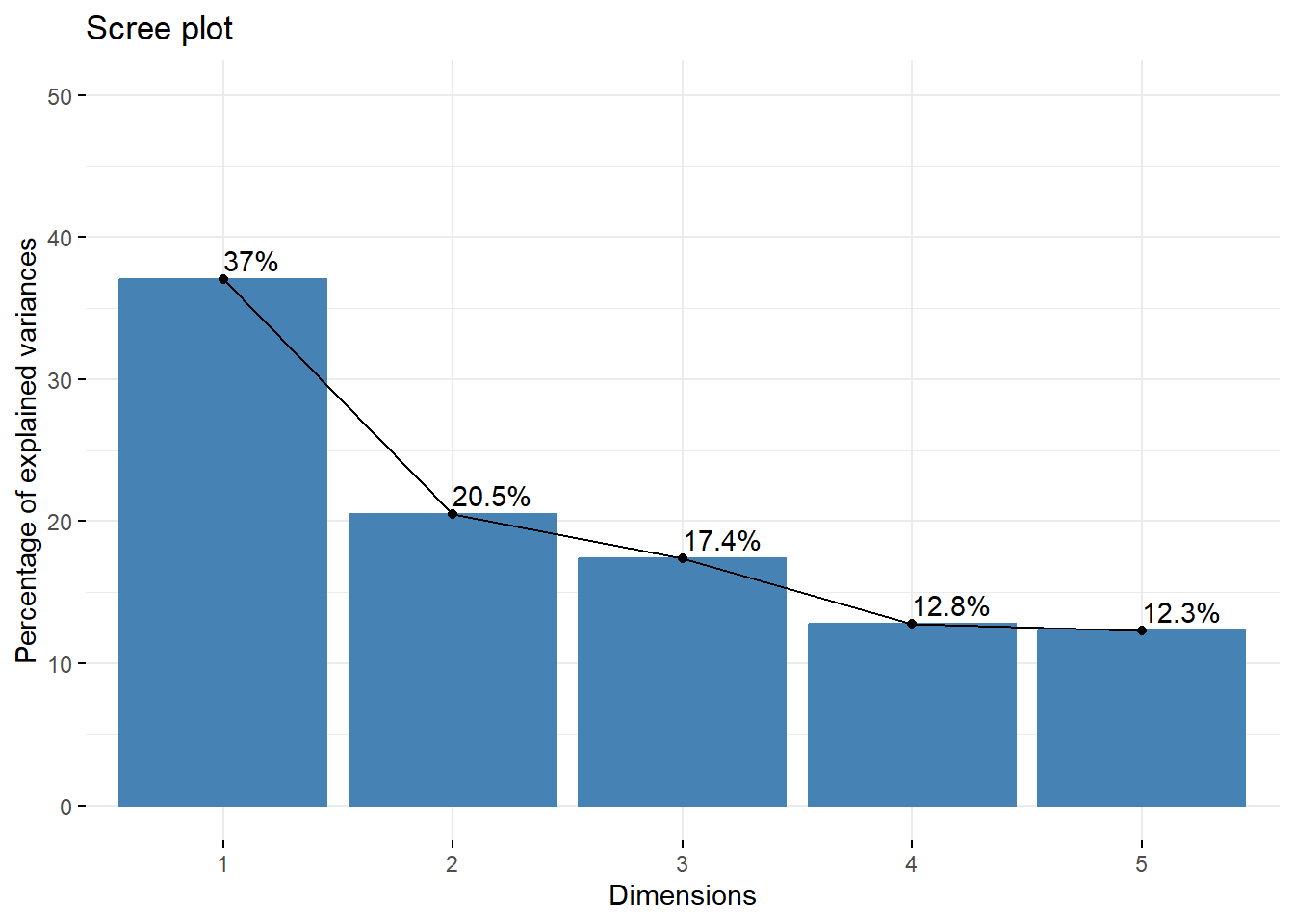
kable(acp$co, digits = 3)| Comp1 | Comp2 | Comp3 | |
|---|---|---|---|
| mate | -0.706 | 0.259 | 0.204 |
| cien | -0.612 | 0.242 | 0.602 |
| soci | -0.645 | -0.420 | -0.351 |
| text | -0.648 | -0.496 | -0.105 |
| imag | -0.378 | 0.691 | -0.576 |
# exam como ilustrativa
Gexam <- cor(admi$exam, acp$li)
rownames(Gexam)<-"exam"
kable(Gexam, digits = 3)| Axis1 | Axis2 | Axis3 | |
|---|---|---|---|
| exam | -0.985 | 0.159 | 0.023 |
s.corcircle(acp$co)
s.arrow(Gexam, add.plot = TRUE, boxes = FALSE)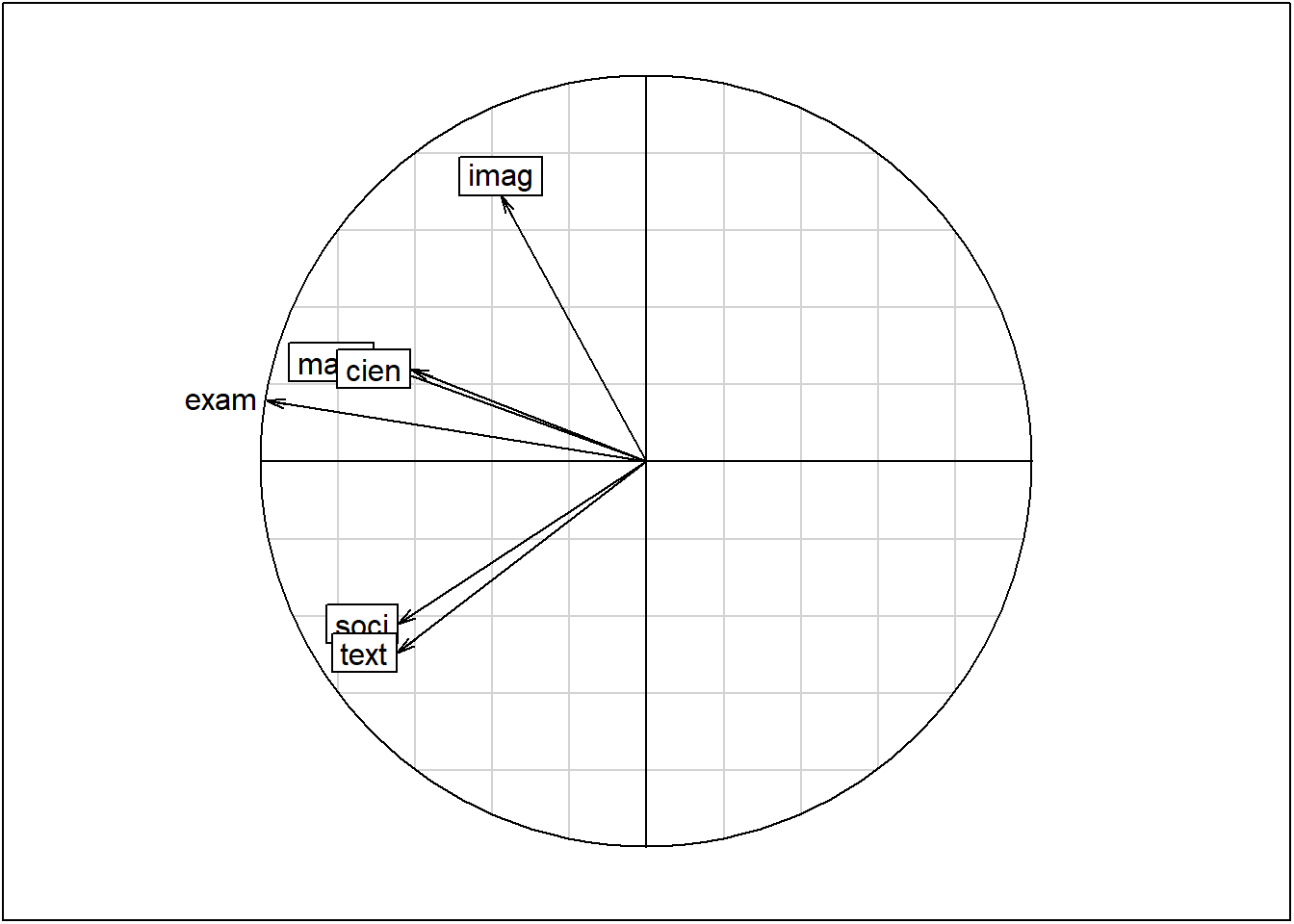
# Plano con los ejes 2 y 3
s.corcircle(acp$co, xax = 2, yax = 3)
s.arrow(Gexam, xax = 2, yax = 3,
add.plot = TRUE, boxes = FALSE)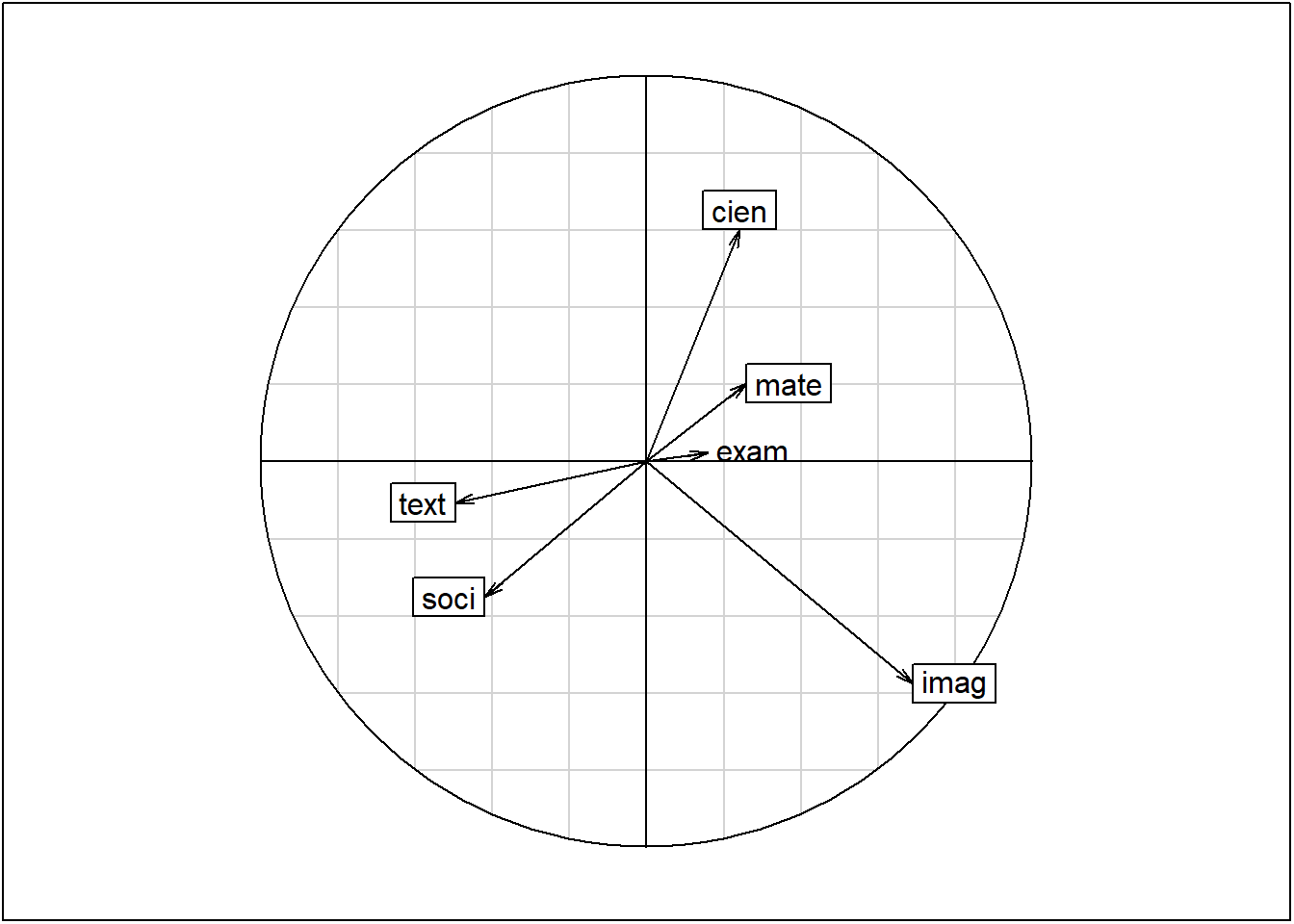
# Código experimental
G_df = as.data.frame(acp$co)
fig <- plot_ly(G_df, x = ~Comp1, y = ~Comp2, z = ~Comp3)
# Graficare como puntos pero sería mejor como flechas
fig <- fig %>% add_markers()
fig <- fig %>% add_text(text = rownames(G_df))
fig <- fig %>% add_markers(x = 0, y = 0, z = 0)
sphere <- function(n = 100){
theta <- seq(0, pi, length.out = n)
phi <- seq(0, 2 * pi, length.out = n)
r <- 1
x <- r * outer(sin(theta), cos(phi))
y <- r * outer(sin(theta), sin(phi))
z <- r * outer(cos(theta), rep(1, length(phi)))
return(list(x = x, y = y, z = z))
}
s_c <- sphere()
fig <- fig %>% add_trace(x = s_c$x, y = s_c$y, z = s_c$z,
type = 'surface', opacity = 0.2,
colorscale = "Greys", # cambiar por valor fijo
showscale = FALSE)
fig <- fig %>% layout(title = "vectores columna\n(variables)",
scene = list(xaxis = list(range = c(-1,1)),
yaxis = list(range = c(-1,1)),
zaxis = list(range = c(-1,1)),
aspectratio = list(x=1,y=1,z=1),
camera = list(eye = list(x=1,y=1,z=1))),
showlegend = FALSE)
figAyudas numéricas para interpretación (y gráficos asociados):
Código
var <- get_pca_var(acp)
varPrincipal Component Analysis Results for variables
===================================================
Name Description
1 "$coord" "Coordinates for the variables"
2 "$cor" "Correlations between variables and dimensions"
3 "$cos2" "Cos2 for the variables"
4 "$contrib" "contributions of the variables" Código
fviz_contrib(acp, choice = "var", axes = 3)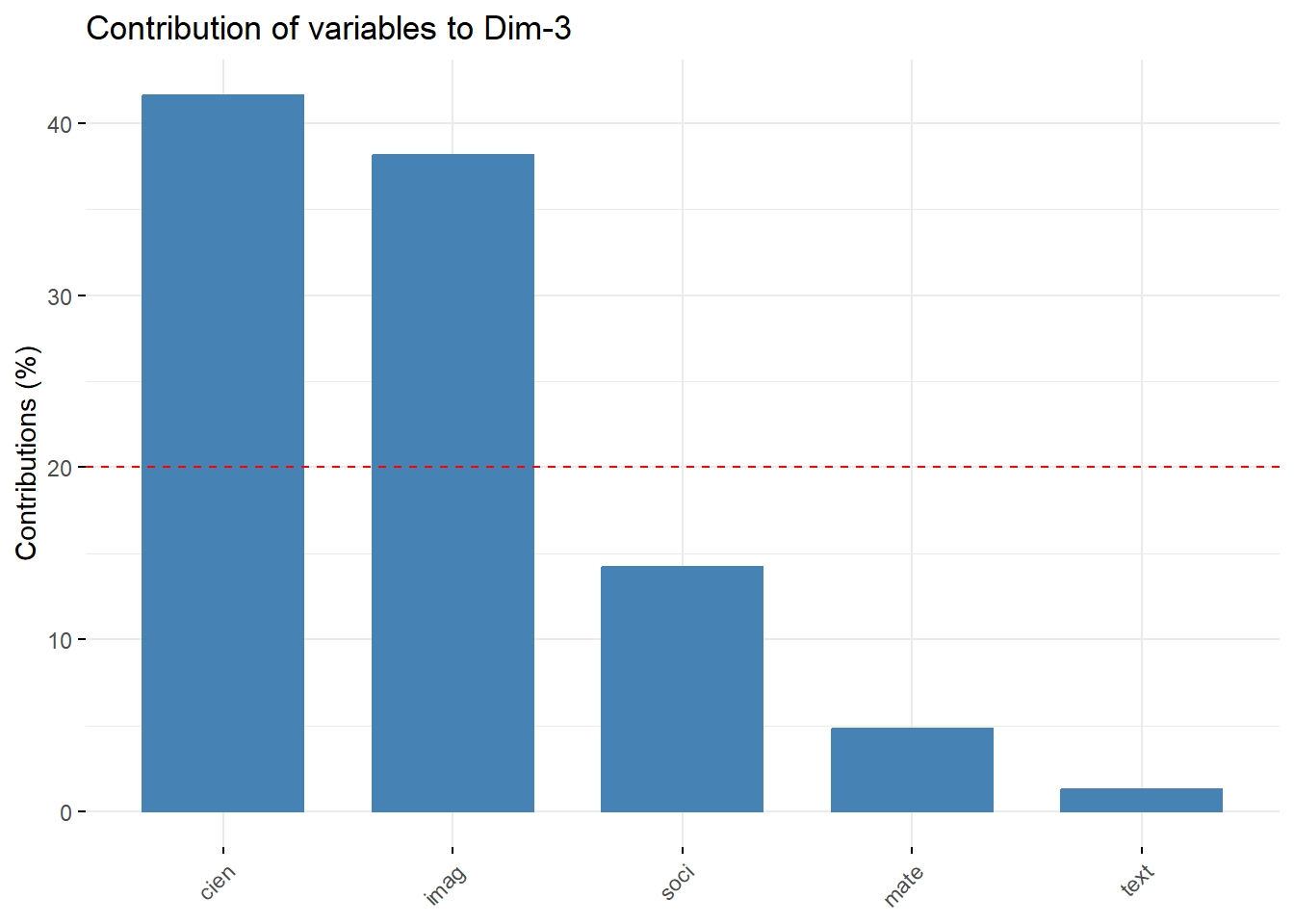
Código
fviz_pca_var(acp, axes=c(3,2), col.var="cos2",
gradient.cols = c("darkred",
"darkorange",
"darkgreen"))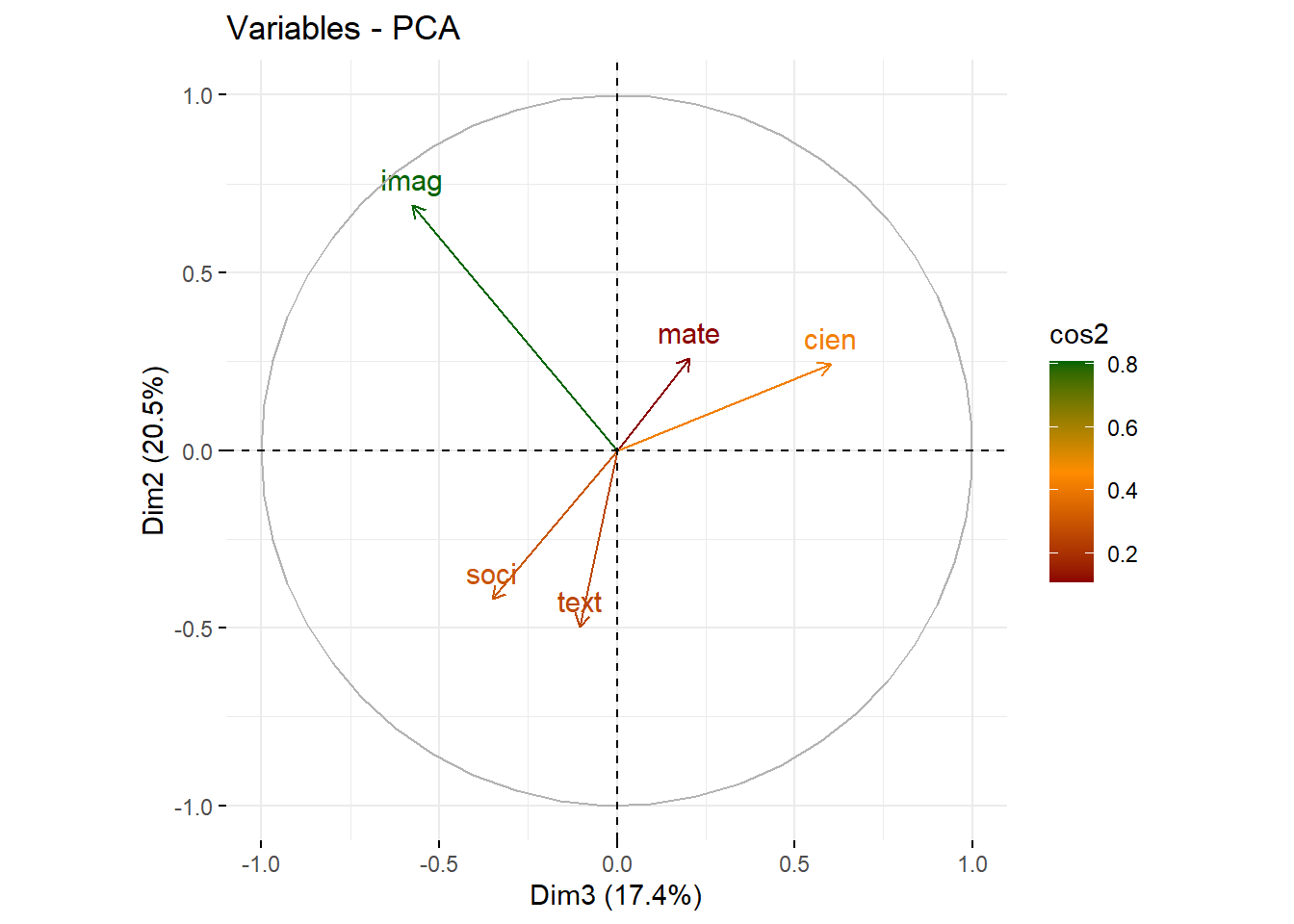
Elementos suplementarios:
Ysupcat <- admi[, c(1, 8:13)]
sup <- supqual(acp, Ysupcat)
kable(cbind(wcat = sup$wcat, d2 = sup$dis2, sup$coor),
digits = 3)| wcat | d2 | Axis1 | Axis2 | Axis3 | |
|---|---|---|---|---|---|
| Biol | 0.142 | 0.117 | -0.036 | -0.152 | -0.080 |
| Esta | 0.148 | 0.403 | 0.601 | 0.082 | -0.109 |
| Farm | 0.164 | 0.346 | 0.427 | -0.315 | 0.068 |
| Fisi | 0.184 | 0.214 | -0.416 | 0.114 | 0.108 |
| Geol | 0.101 | 0.244 | -0.483 | -0.010 | -0.071 |
| Mate | 0.119 | 0.751 | -0.573 | 0.496 | -0.014 |
| Quim | 0.142 | 0.106 | 0.280 | -0.127 | 0.036 |
| F | 0.288 | 0.229 | 0.410 | -0.211 | 0.117 |
| M | 0.712 | 0.037 | -0.166 | 0.085 | -0.047 |
| bajo | 0.402 | 0.267 | 0.506 | 0.088 | 0.035 |
| medio | 0.416 | 0.021 | -0.099 | -0.030 | -0.068 |
| alto | 0.182 | 0.878 | -0.891 | -0.125 | 0.078 |
| Bogo | 0.699 | 0.040 | -0.182 | 0.053 | -0.062 |
| Cund | 0.085 | 0.080 | 0.039 | -0.246 | -0.025 |
| Otro | 0.216 | 0.382 | 0.573 | -0.075 | 0.210 |
| a16m | 0.265 | 0.052 | 0.057 | -0.079 | 0.195 |
| a17 | 0.384 | 0.079 | -0.220 | 0.044 | -0.118 |
| a18 | 0.126 | 0.162 | 0.372 | -0.117 | -0.068 |
| a19M | 0.225 | 0.059 | 0.102 | 0.084 | 0.011 |
| siLE | 0.103 | 3.636 | 1.698 | 0.654 | 0.388 |
| noLE | 0.897 | 0.048 | -0.196 | -0.075 | -0.045 |
| siMa | 0.708 | 0.309 | 0.460 | -0.116 | -0.119 |
| noMa | 0.292 | 1.814 | -1.115 | 0.281 | 0.287 |
| Axis1 | Axis2 | Axis3 | Axis1 | Axis2 | Axis3 | |
|---|---|---|---|---|---|---|
| Biol | -0.228 | -1.287 | -0.730 | 0.011 | 0.198 | 0.054 |
| Esta | 3.885 | 0.711 | -1.024 | 0.897 | 0.017 | 0.029 |
| Farm | 2.928 | -2.908 | 0.684 | 0.526 | 0.287 | 0.013 |
| Fisi | -3.064 | 1.127 | 1.160 | 0.812 | 0.061 | 0.055 |
| Geol | -2.507 | -0.071 | -0.540 | 0.956 | 0.000 | 0.021 |
| Mate | -3.261 | 3.799 | -0.114 | 0.437 | 0.328 | 0.000 |
| Quim | 1.763 | -1.071 | 0.331 | 0.745 | 0.152 | 0.012 |
| F | 4.036 | -2.793 | 1.675 | 0.733 | 0.194 | 0.059 |
| M | -4.036 | 2.793 | -1.675 | 0.733 | 0.194 | 0.059 |
| bajo | 6.430 | 1.496 | 0.642 | 0.961 | 0.029 | 0.005 |
| medio | -1.300 | -0.530 | -1.292 | 0.468 | 0.043 | 0.217 |
| alto | -6.512 | -1.225 | 0.834 | 0.905 | 0.018 | 0.007 |
| Bogo | -4.286 | 1.685 | -2.129 | 0.828 | 0.071 | 0.096 |
| Cund | 0.186 | -1.567 | -0.174 | 0.019 | 0.755 | 0.008 |
| Otro | 4.654 | -0.814 | 2.493 | 0.860 | 0.015 | 0.116 |
| a16m | 0.526 | -0.989 | 2.649 | 0.062 | 0.121 | 0.738 |
| a17 | -2.697 | 0.717 | -2.115 | 0.616 | 0.024 | 0.178 |
| a18 | 2.188 | -0.921 | -0.585 | 0.855 | 0.084 | 0.029 |
| a19M | 0.847 | 0.942 | 0.128 | 0.176 | 0.120 | 0.002 |
| siLE | 8.927 | 4.624 | 2.973 | 0.793 | 0.118 | 0.041 |
| noLE | -8.927 | -4.624 | -2.973 | 0.793 | 0.118 | 0.041 |
| siMa | 11.093 | -3.763 | -4.171 | 0.685 | 0.044 | 0.046 |
| noMa | -11.093 | 3.763 | 4.171 | 0.685 | 0.044 | 0.046 |
plot(acp, Tcol = FALSE, ucal = 100, cex.row = 0.2,
xlim = c(-1,1.5), ylim = c(-0.6,0.6))
points(sup$coor, col = 2, pch = 16)
text(sup$coor, labels = rownames(sup$coor),
col = 2, pos = 1, font = 3)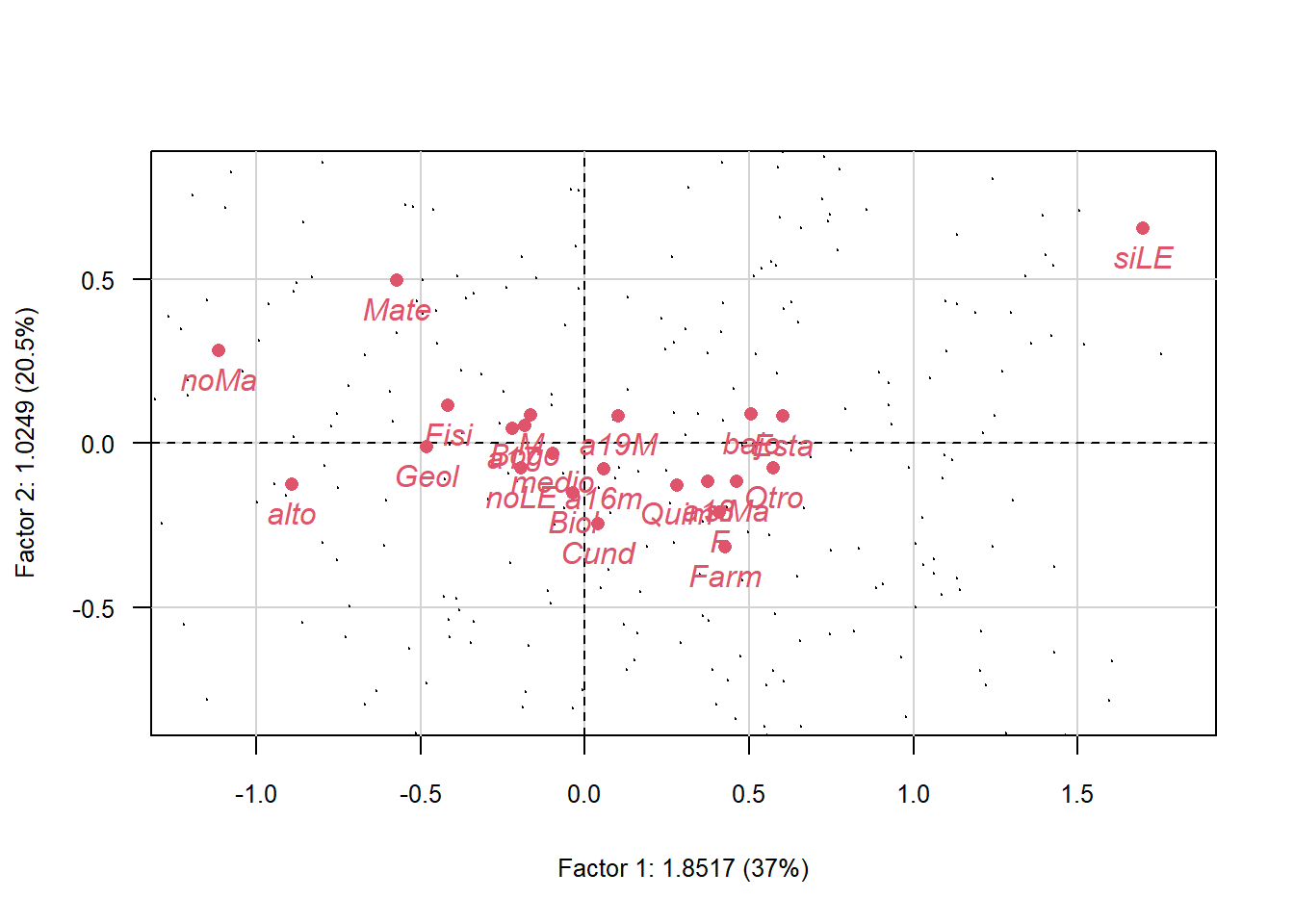
plot(acp, Tcol = FALSE, ucal = 100, cex.row = 0.2,
xlim = c(-1,1.5), ylim = c(-0.6,0.6))
s.arrow(acp$co, add.plot = TRUE, boxes = FALSE)
s.arrow(Gexam, add.plot = TRUE, boxes = FALSE)
points(sup$coor[8:9,], col = 3, pch = 16)
text(sup$coor[8:9,], labels = rownames(sup$coor[8:9,]),
col = 3, pos = 1, font = 3)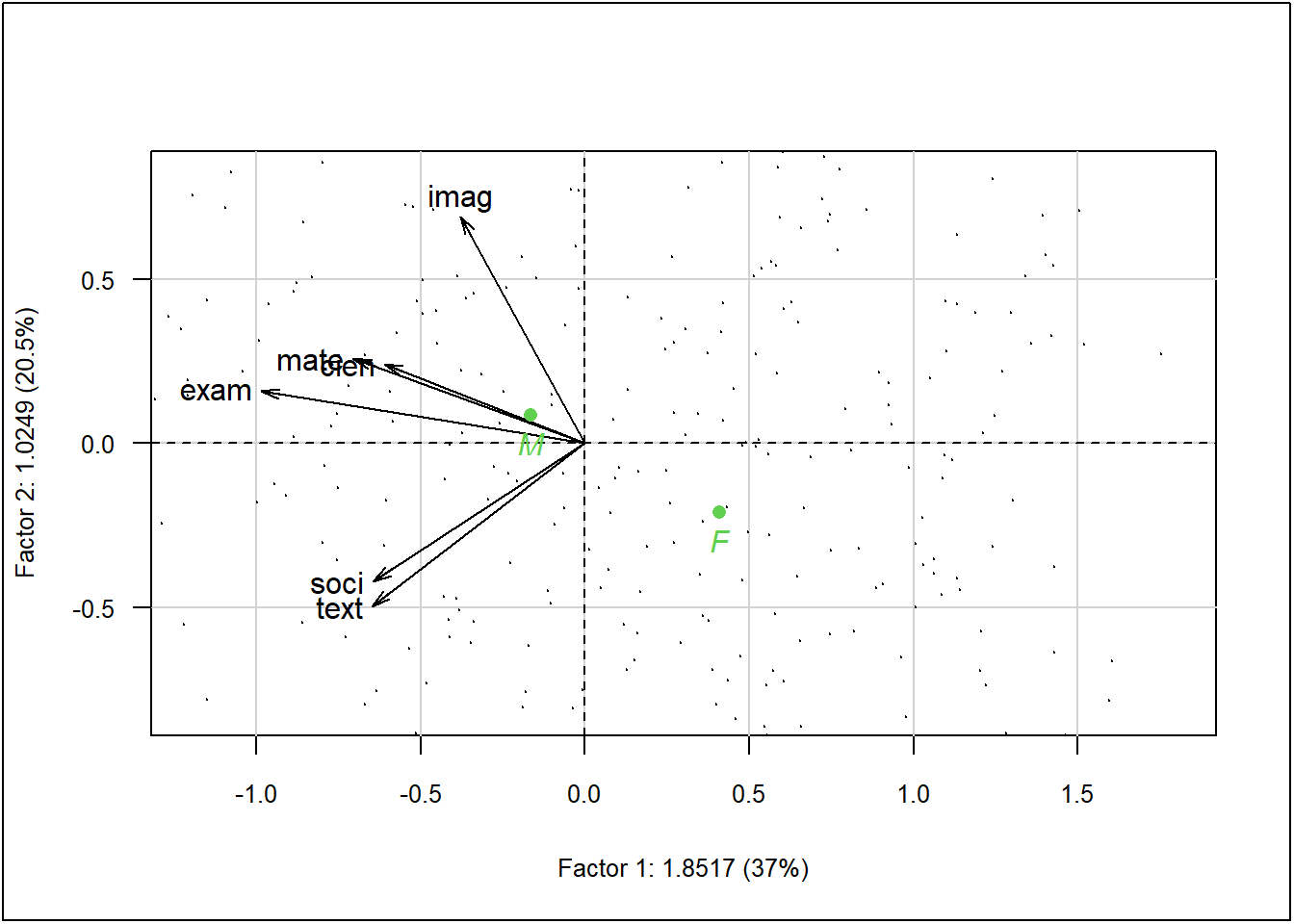
plot(acp, Tcol = FALSE, ucal = 100, cex.row = 0.2,
xlim = c(-1,1.5), ylim = c(-0.6,0.6))
s.arrow(acp$co, add.plot = TRUE, boxes = FALSE)
s.arrow(Gexam, add.plot = TRUE, boxes = FALSE)
points(sup$coor[16:19,], col = 5, pch = 16)
text(sup$coor[16:19,], labels = rownames(sup$coor[16:19,]),
col = 5, pos = 1, font = 3)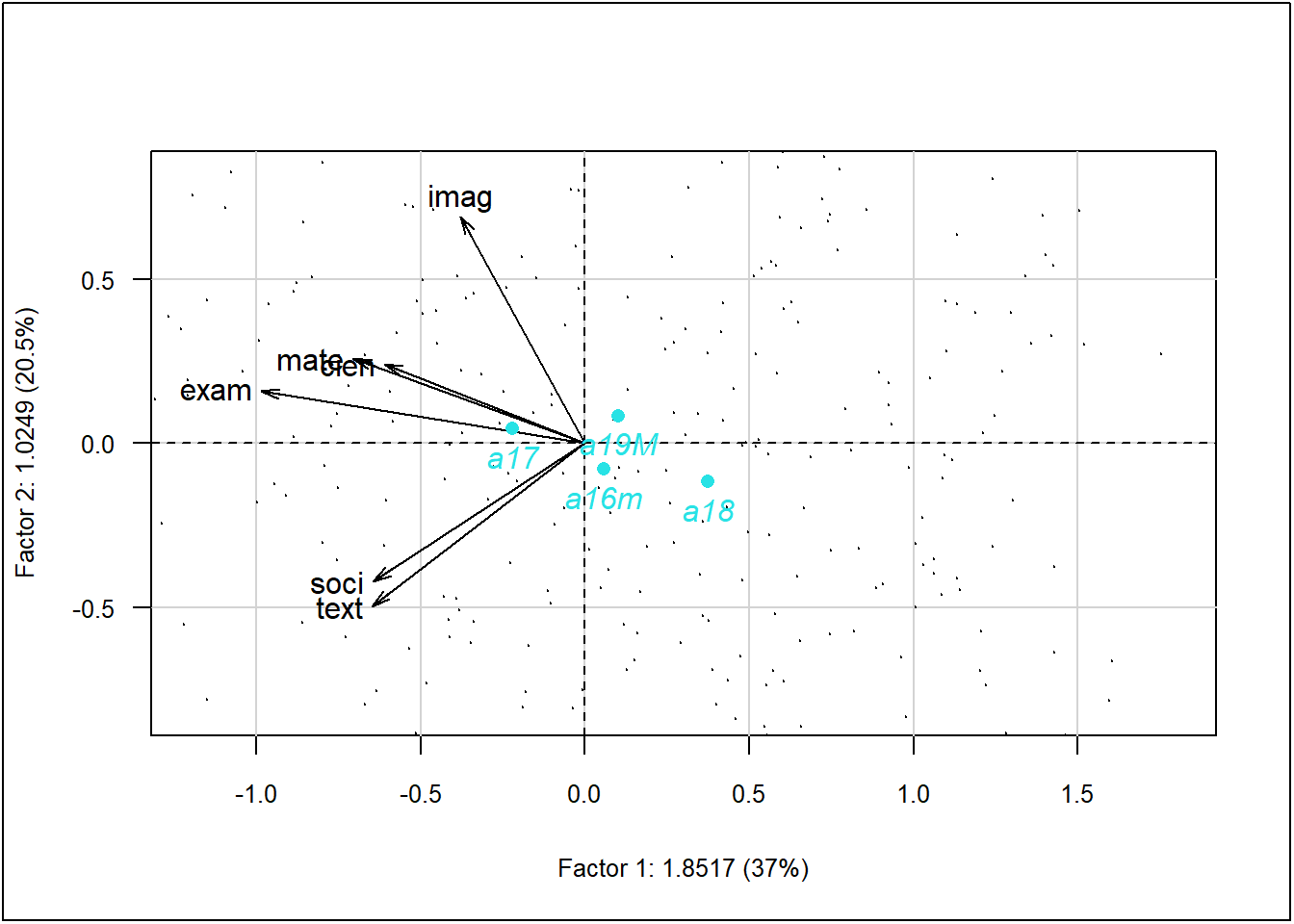
plot(acp, Tcol = FALSE, ucal = 100, cex.row = 0.2,
xlim = c(-1,1.5), ylim = c(-0.6,0.6))
s.arrow(acp$co, add.plot = TRUE, boxes = FALSE)
s.arrow(Gexam, add.plot = TRUE, boxes = FALSE)
points(sup$coor[1:7,], col = 6, pch = 16)
text(sup$coor[1:7,], labels = rownames(sup$coor[1:7,]),
col = 6, pos = 1, font = 3)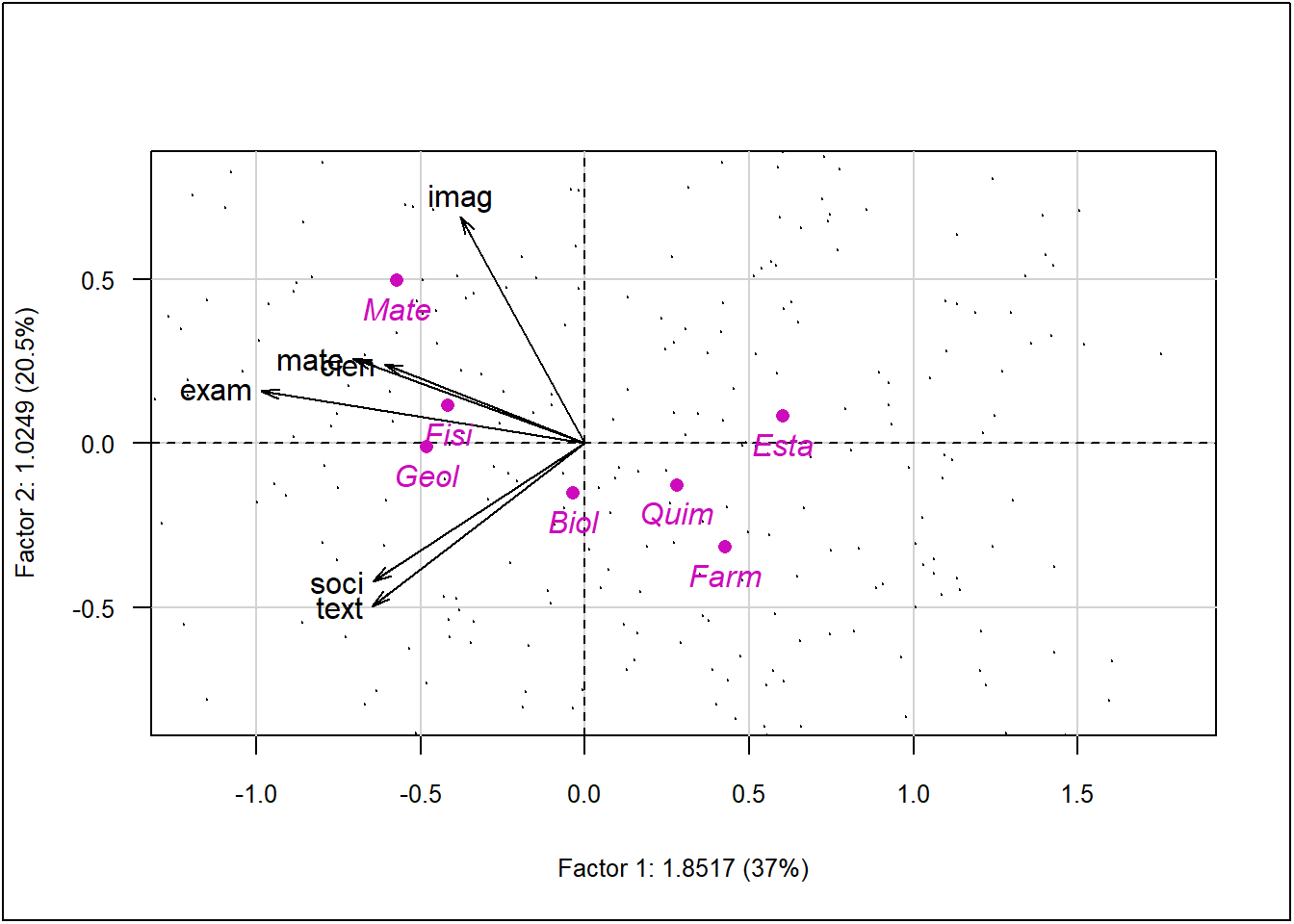
plot(sup$coor[1:7,2:3], col = 6, pch = 16,
xlim = c(-1,1), ylim = c(-1,1),
bty="n", axes = FALSE)
text(sup$coor[1:7,2:3], labels = rownames(sup$coor[1:7,]),
col = 6, pos = 1, font = 3)
curve(sqrt(1 - x*x), -1, 1, 1e4, add = TRUE, lty = 3)
curve(-sqrt(1 - x*x), -1, 1, 1e4, add = TRUE, lty = 3)
abline(h=0, col="grey"); abline(v=0, col="grey")
s.arrow(acp$co, add.plot = TRUE, boxes = FALSE,
xax = 2, yax = 3)
s.arrow(Gexam, add.plot = TRUE, boxes = FALSE,
xax = 2, yax = 3)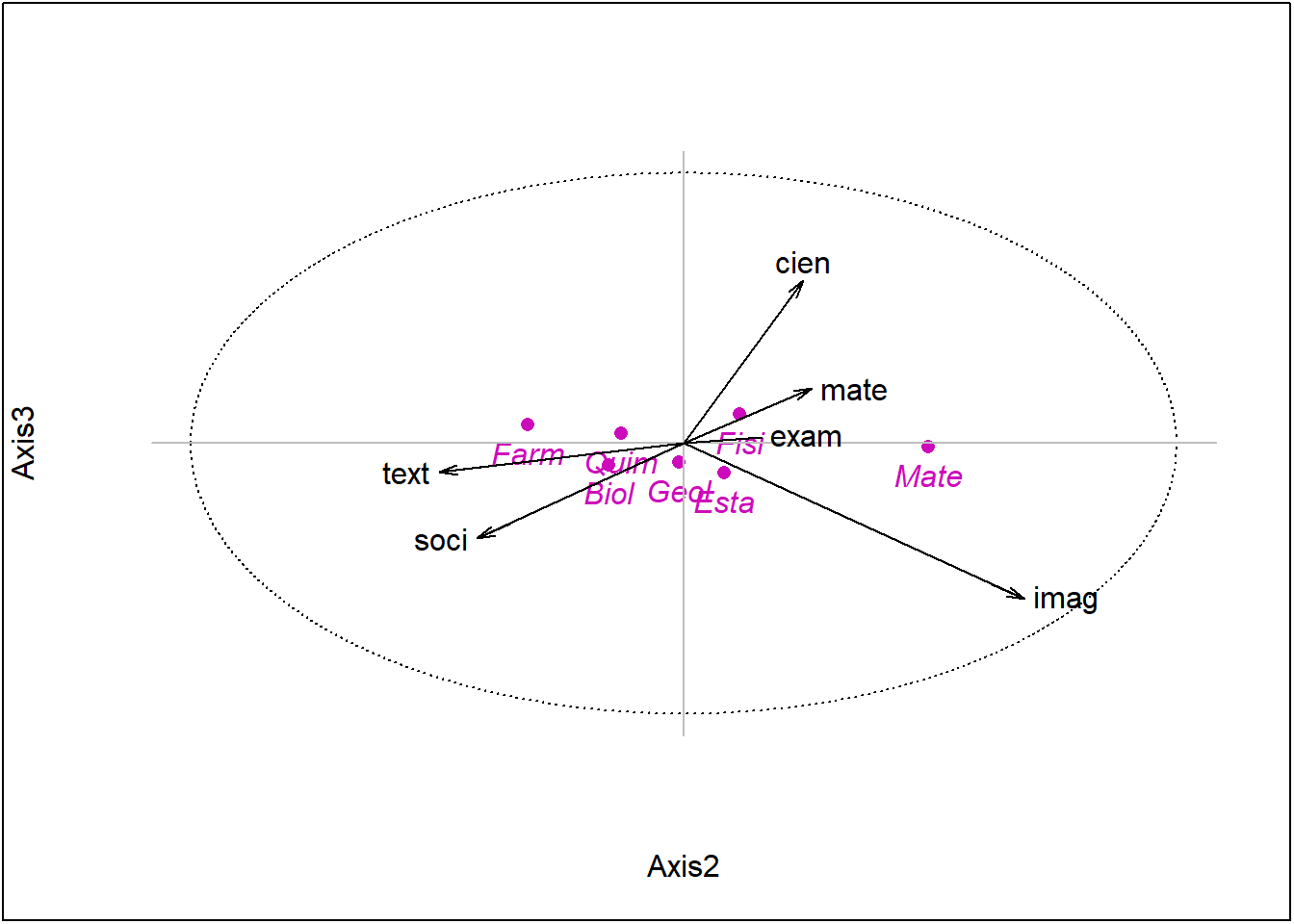
plot(acp, Tcol = FALSE, ucal = 100, cex.row = 0.2)
ade4::s.class(acp$li, admi[, 1], col = rainbow(8)[2:8],
add.plot = TRUE, clabel = 0.5)
plot(acp, Tcol = FALSE, ucal = 100, cex.row = 0.2,
xlim = c(-1,1.5), ylim = c(-0.6,0.6))
s.arrow(acp$co, add.plot = TRUE, boxes = FALSE)
s.arrow(Gexam, add.plot = TRUE, boxes = FALSE)
points(sup$coor[10:12,], col = 4, pch = 16)
text(sup$coor[10:12,], labels = rownames(sup$coor[10:12,]),
col = 4, pos = 1, font = 3)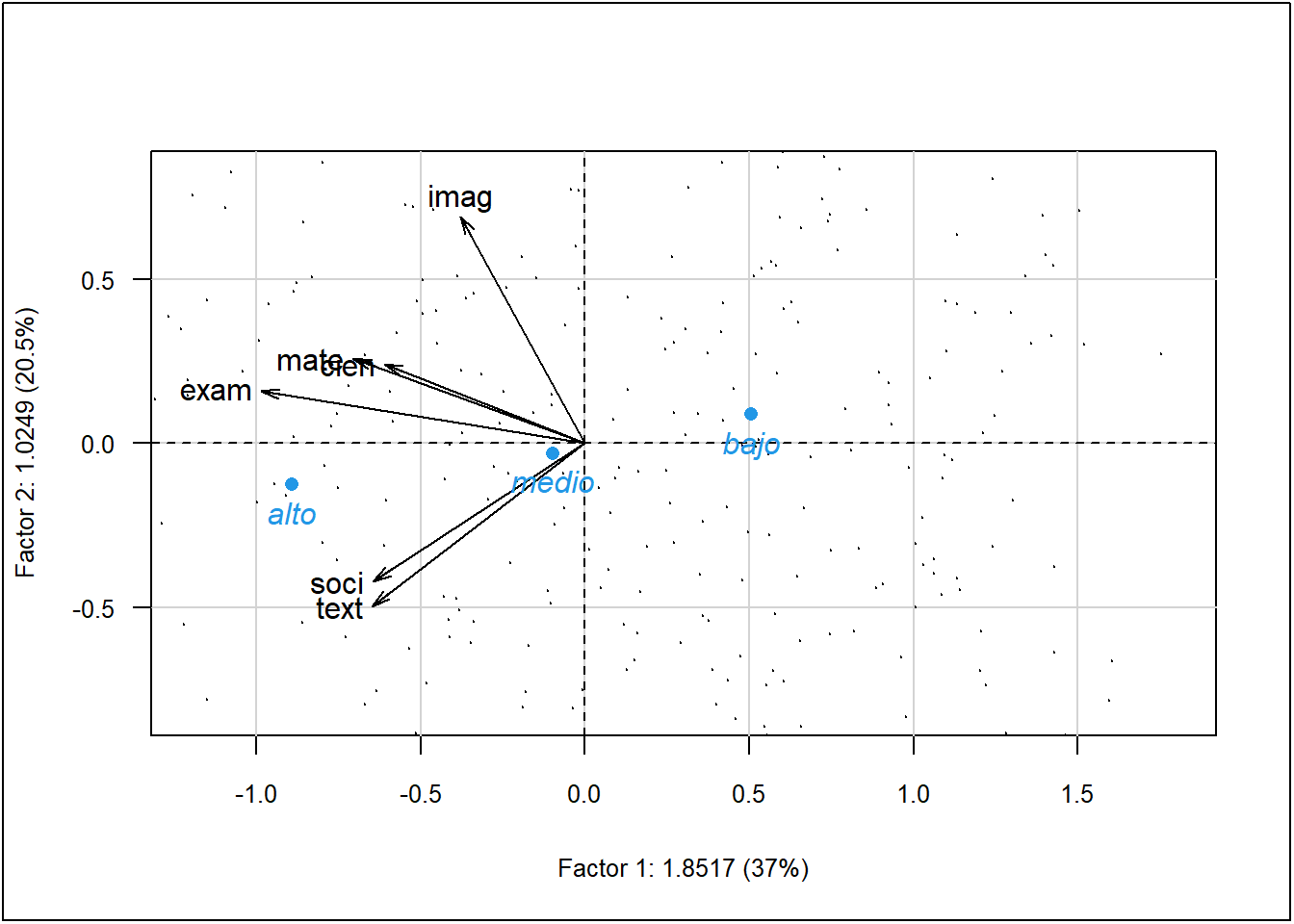
fviz_pca_biplot(acp, label = "var",
habillage = admi$estr,
addEllipses = TRUE)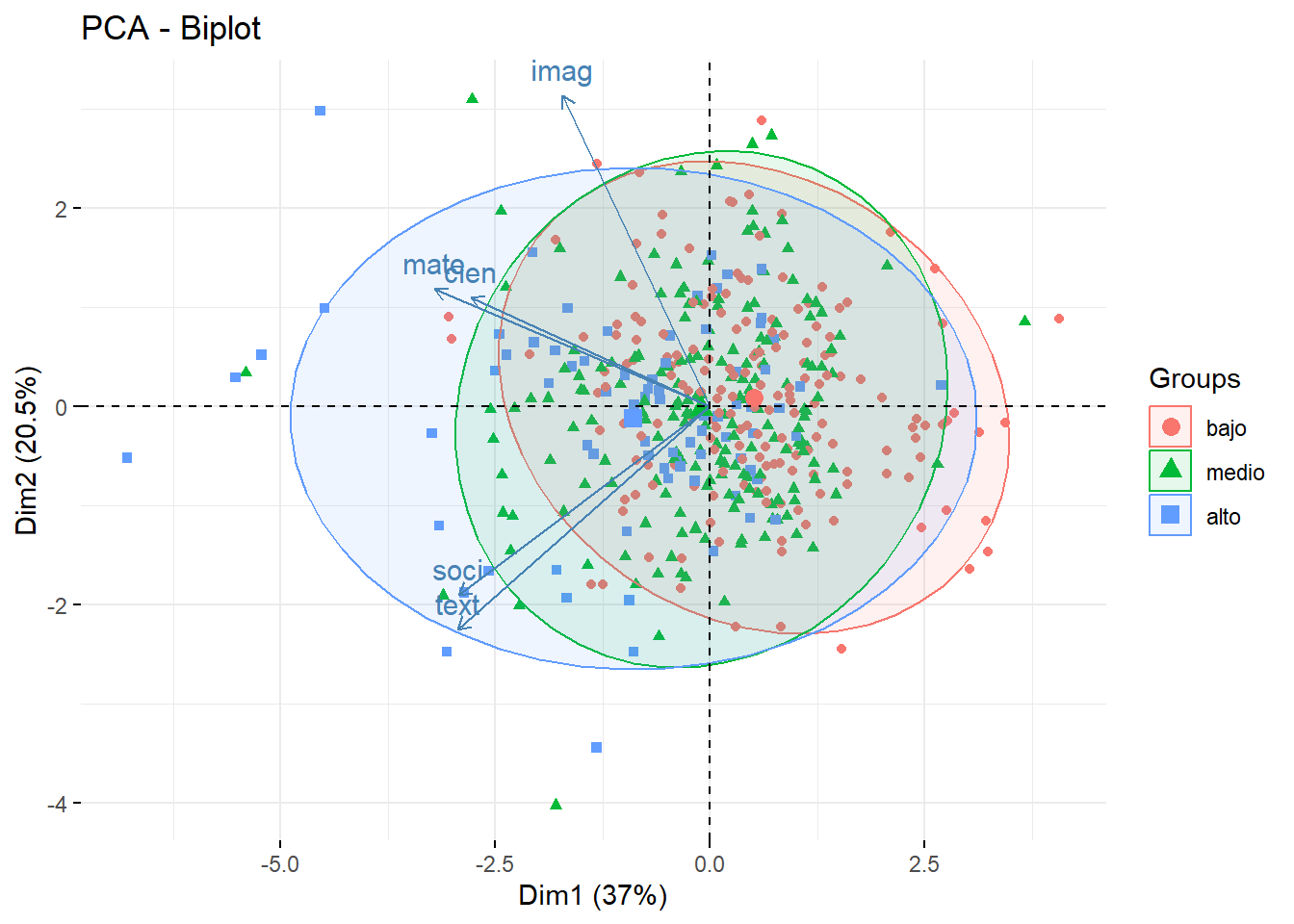
fviz_pca_biplot(acp, axes = c(3,2),
label = "var",
habillage = admi$estr,
addEllipses = TRUE)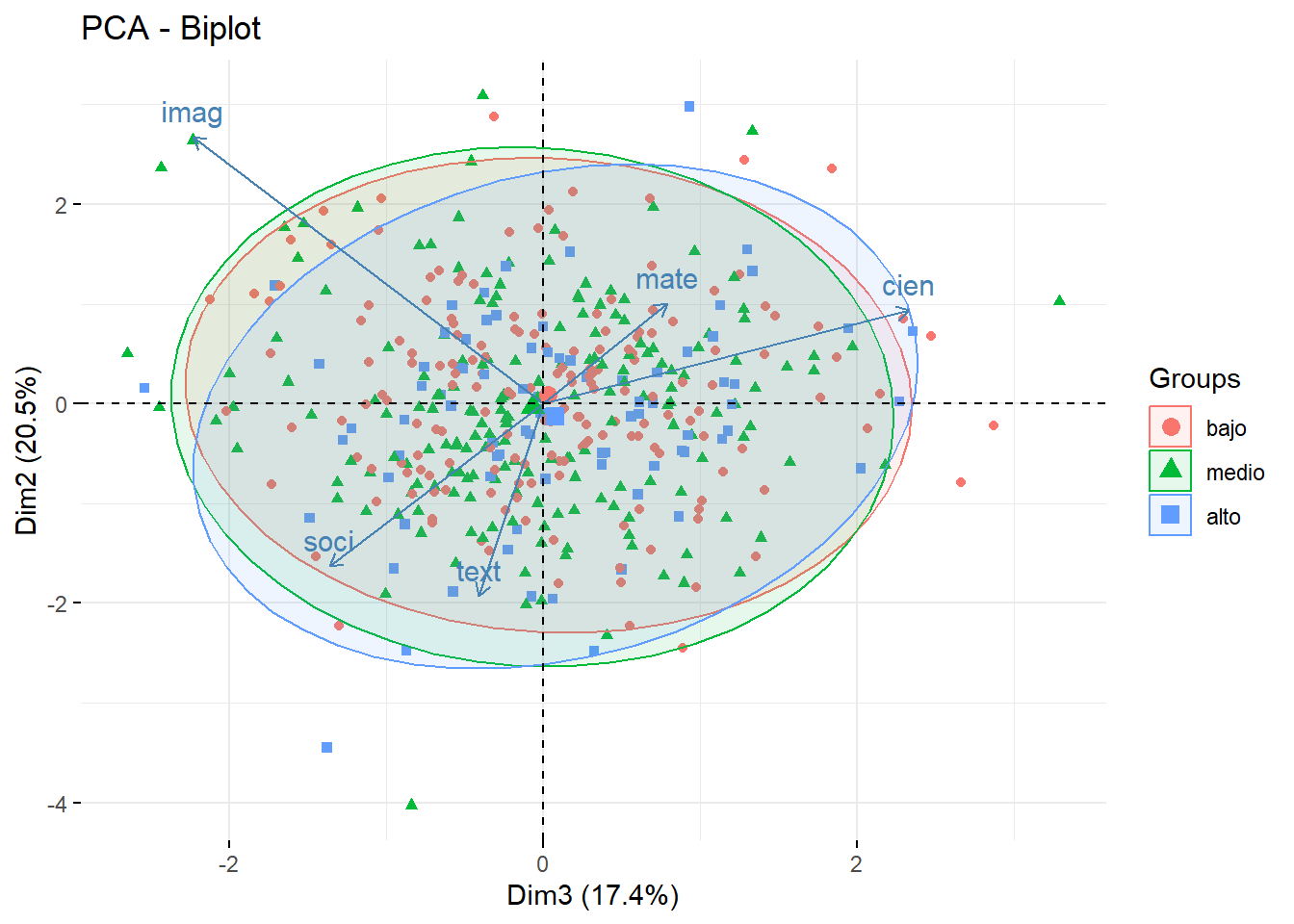
2.5 Taller (ACP de whisky)
price malt aging taste
Min. : 55.0 Min. : 20.0 Min. : 5.00 Min. :0.00
1st Qu.: 73.0 1st Qu.: 30.0 1st Qu.: 8.00 1st Qu.:2.00
Median : 83.0 Median : 40.0 Median :10.00 Median :2.00
Mean : 85.7 Mean : 47.4 Mean : 9.53 Mean :2.23
3rd Qu.: 91.5 3rd Qu.: 45.0 3rd Qu.:12.00 3rd Qu.:3.00
Max. :160.0 Max. :100.0 Max. :12.50 Max. :4.00 summary(Whisky[, 3]) low med pure
11 17 7 cov(data) # Muestral price malt aging taste
price 395.563 359.735 24.8908 7.6261
malt 359.735 758.247 27.6941 8.7882
aging 24.891 27.694 6.7197 0.9345
taste 7.626 8.788 0.9345 1.4756cor(data) price malt aging taste
price 1.0000 0.6569 0.4828 0.3156
malt 0.6569 1.0000 0.3880 0.2627
aging 0.4828 0.3880 1.0000 0.2968
taste 0.3156 0.2627 0.2968 1.0000acp <- dudi.pca(data, scannf = FALSE, nf = n)
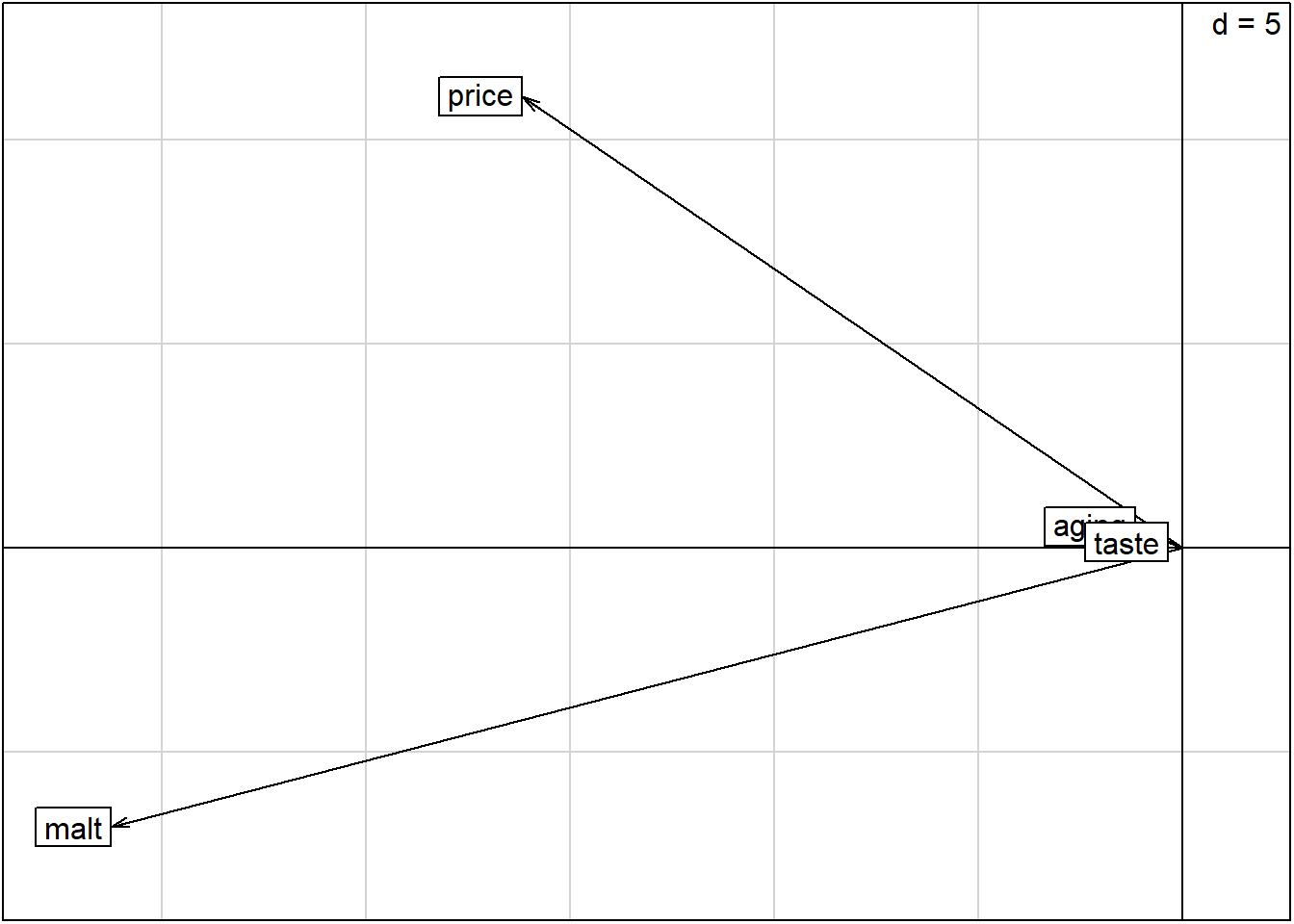
s.corcircle(acp$co)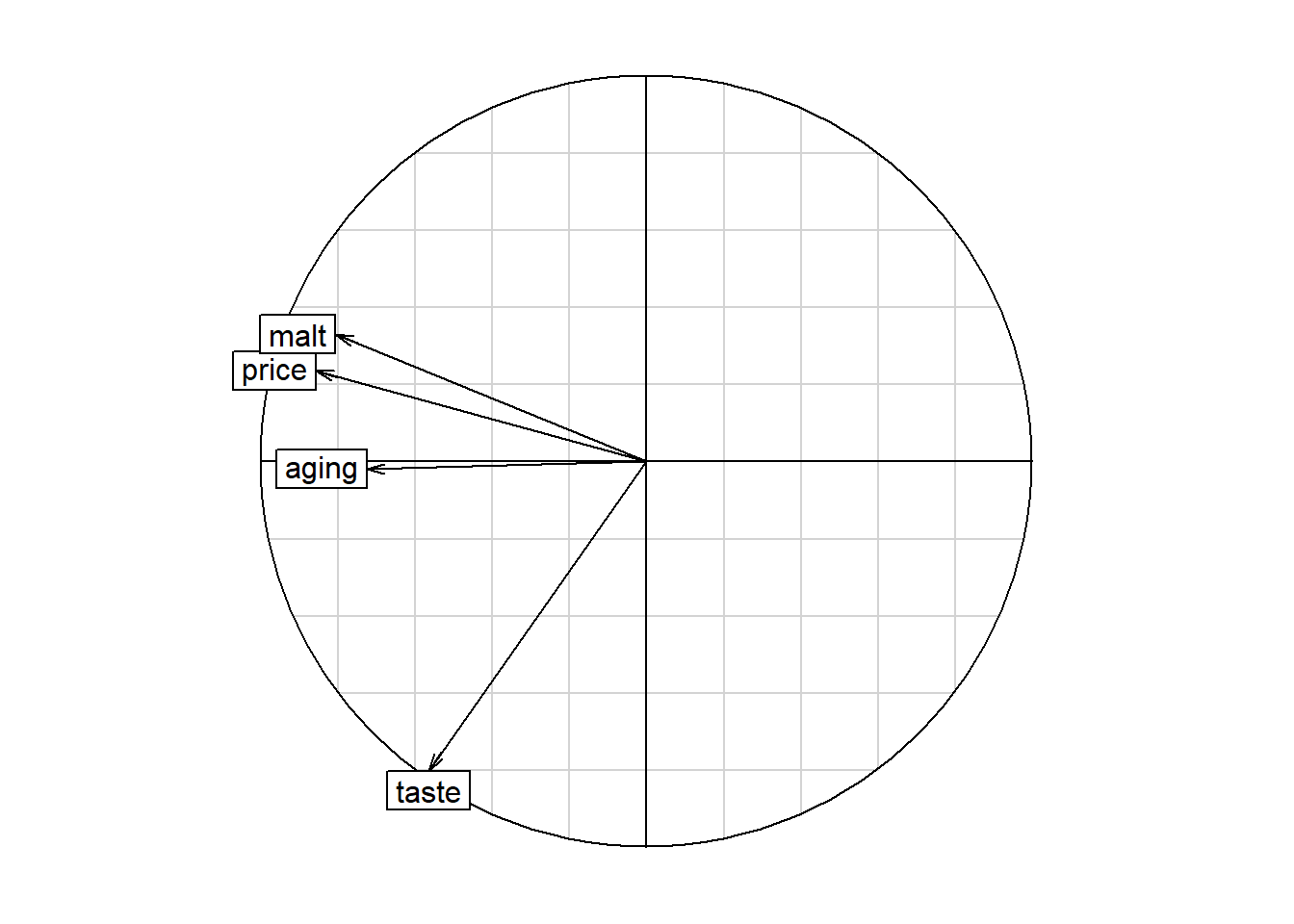
inertia_acp <- inertia(acp, row.inertia = TRUE, col.inertia = TRUE)
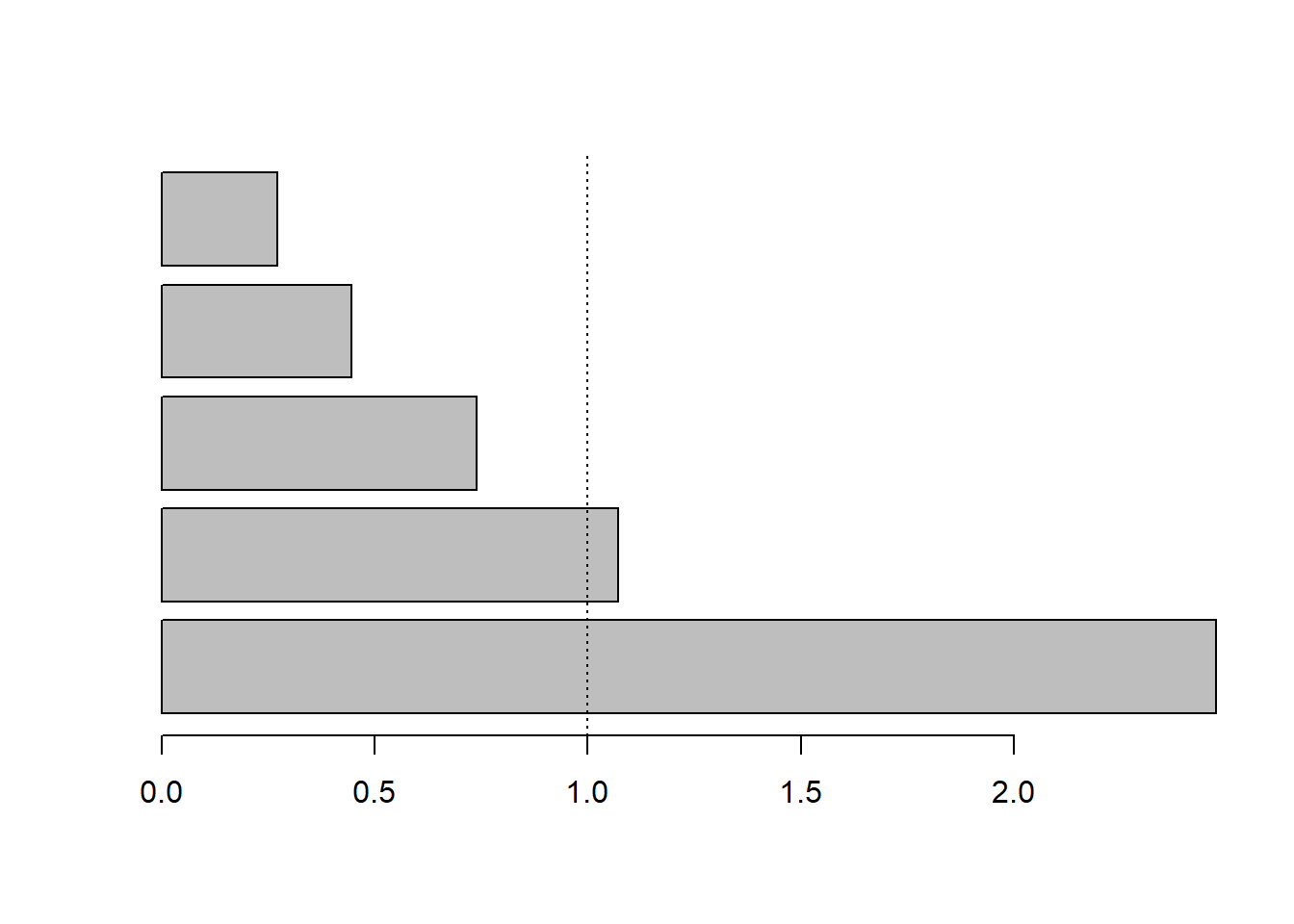
valp <- t(inertia_acp$tot.inertia) # valores propios
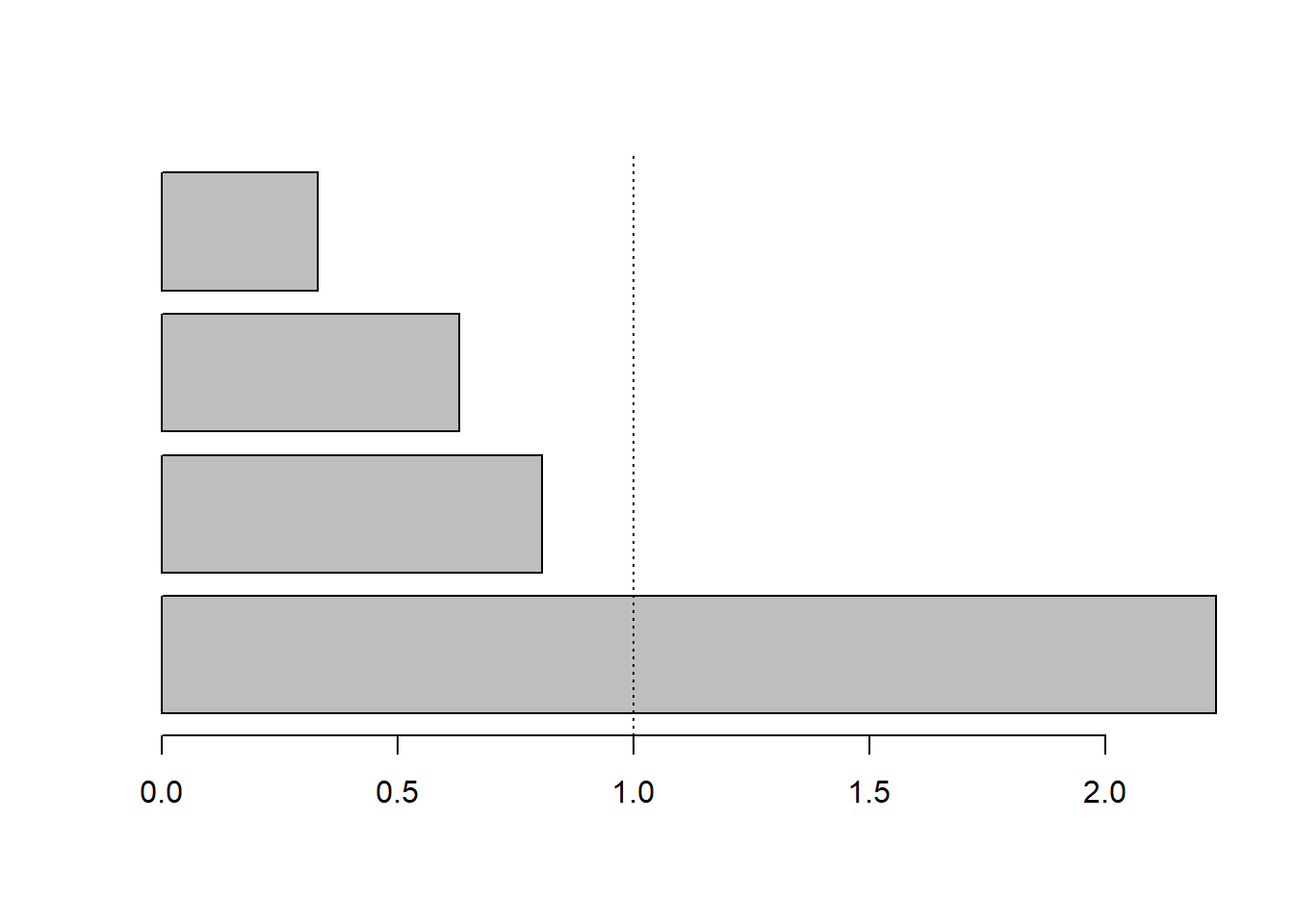
kable(valp, digits = 2)| Ax1 | Ax2 | Ax3 | Ax4 | |
|---|---|---|---|---|
| inertia | 2.23 | 0.81 | 0.63 | 0.33 |
| cum | 2.23 | 3.04 | 3.67 | 4.00 |
| cum(%) | 55.83 | 75.99 | 91.73 | 100.00 |
kable(inertia_acp$col.abs, digits = 1)| Axis1 | Axis2 | Axis3 | Axis4 | |
|---|---|---|---|---|
| price | 33.0 | 6.8 | 3.1 | 57.1 |
| malt | 29.1 | 13.5 | 17.2 | 40.2 |
| aging | 23.6 | 0.0 | 73.7 | 2.6 |
| taste | 14.3 | 79.6 | 6.0 | 0.1 |
kable(inertia_acp$col.rel, digits = 1)| Axis1 | Axis2 | Axis3 | Axis4 | |
|---|---|---|---|---|
| price | -73.7 | 5.5 | 1.9 | 18.9 |
| malt | -65.0 | 10.9 | 10.8 | -13.3 |
| aging | -52.7 | 0.0 | -46.4 | -0.9 |
| taste | -32.0 | -64.2 | 3.7 | 0.0 |
kable(inertia_acp$row.abs, digits = 1)| Axis1 | Axis2 | Axis3 | Axis4 |
|---|---|---|---|
| 3.4 | 4.4 | 5.7 | 0.8 |
| 6.4 | 0.9 | 3.1 | 0.0 |
| 3.3 | 0.7 | 0.0 | 0.0 |
| 0.2 | 0.3 | 8.0 | 0.1 |
| 0.0 | 4.4 | 6.1 | 0.6 |
| 6.7 | 5.9 | 2.1 | 0.6 |
| 4.2 | 5.3 | 0.5 | 0.0 |
| 6.1 | 0.7 | 4.0 | 2.0 |
| 5.5 | 6.4 | 1.4 | 1.3 |
| 0.2 | 8.0 | 10.0 | 3.1 |
| 0.0 | 0.1 | 6.3 | 0.1 |
| 3.4 | 1.2 | 0.9 | 0.0 |
| 1.4 | 4.1 | 0.3 | 2.2 |
| 0.3 | 2.1 | 3.5 | 0.4 |
| 0.3 | 0.0 | 1.0 | 0.0 |
| 0.1 | 9.0 | 0.2 | 1.6 |
| 0.2 | 0.0 | 0.2 | 0.7 |
| 0.4 | 0.0 | 0.1 | 0.0 |
| 0.1 | 0.0 | 0.1 | 0.2 |
| 0.1 | 6.9 | 1.7 | 0.5 |
| 0.2 | 0.0 | 0.2 | 0.0 |
| 0.5 | 2.2 | 0.6 | 0.1 |
| 0.3 | 0.0 | 5.7 | 0.2 |
| 2.1 | 0.5 | 1.5 | 7.5 |
| 1.1 | 0.1 | 6.6 | 2.0 |
| 4.1 | 3.6 | 0.3 | 7.0 |
| 1.2 | 0.9 | 1.7 | 0.6 |
| 0.4 | 1.6 | 2.5 | 0.6 |
| 8.8 | 0.0 | 16.1 | 1.2 |
| 19.8 | 4.3 | 2.9 | 18.3 |
| 6.2 | 1.2 | 0.6 | 14.0 |
| 2.7 | 2.7 | 0.0 | 16.1 |
| 4.1 | 0.4 | 3.9 | 4.6 |
| 5.1 | 0.4 | 1.6 | 5.4 |
| 1.5 | 21.7 | 0.7 | 8.0 |
kable(inertia_acp$row.rel, digits = 1)| Axis1 | Axis2 | Axis3 | Axis4 |
|---|---|---|---|
| 50.4 | -23.9 | 24.0 | 1.7 |
| 84.1 | -4.3 | 11.5 | -0.1 |
| 92.4 | -7.5 | 0.0 | 0.0 |
| 7.6 | -4.8 | -87.3 | -0.3 |
| 1.4 | -46.0 | -50.0 | -2.6 |
| 70.1 | 22.5 | 6.4 | 1.0 |
| 66.6 | 30.9 | -2.5 | 0.0 |
| 78.5 | -3.1 | 14.4 | -3.9 |
| 65.4 | 27.5 | 4.7 | 2.3 |
| 2.7 | 45.5 | -44.6 | 7.2 |
| 0.2 | -1.3 | -97.5 | 0.9 |
| 83.3 | 10.5 | 5.9 | 0.2 |
| 41.9 | -45.7 | 2.3 | -10.1 |
| -13.2 | -36.6 | -47.5 | 2.6 |
| 47.7 | -2.5 | -49.5 | -0.2 |
| -1.8 | -90.3 | -1.3 | -6.6 |
| 51.3 | 3.3 | 14.1 | 31.3 |
| 94.8 | 0.0 | 5.1 | 0.2 |
| 53.8 | 3.3 | -21.8 | 21.1 |
| -2.7 | -80.0 | 15.1 | 2.3 |
| 78.5 | 0.0 | -20.7 | -0.8 |
| 33.6 | 52.9 | -12.5 | 1.0 |
| -14.7 | 0.7 | -83.0 | 1.6 |
| -55.2 | -4.7 | -10.9 | 29.2 |
| 34.5 | 0.6 | 55.9 | 9.0 |
| -62.6 | -20.1 | -1.4 | 15.9 |
| -57.3 | -14.8 | -23.4 | 4.6 |
| -22.3 | -33.0 | -39.6 | -5.1 |
| -65.1 | 0.0 | 33.5 | 1.3 |
| -79.6 | 6.3 | 3.3 | 10.9 |
| -69.9 | -4.7 | 2.0 | -23.4 |
| -44.5 | 15.9 | -0.1 | -39.5 |
| -68.2 | 2.2 | 18.3 | -11.3 |
| -78.5 | 2.0 | 7.0 | -12.5 |
| -14.0 | 73.1 | -1.9 | -11.0 |
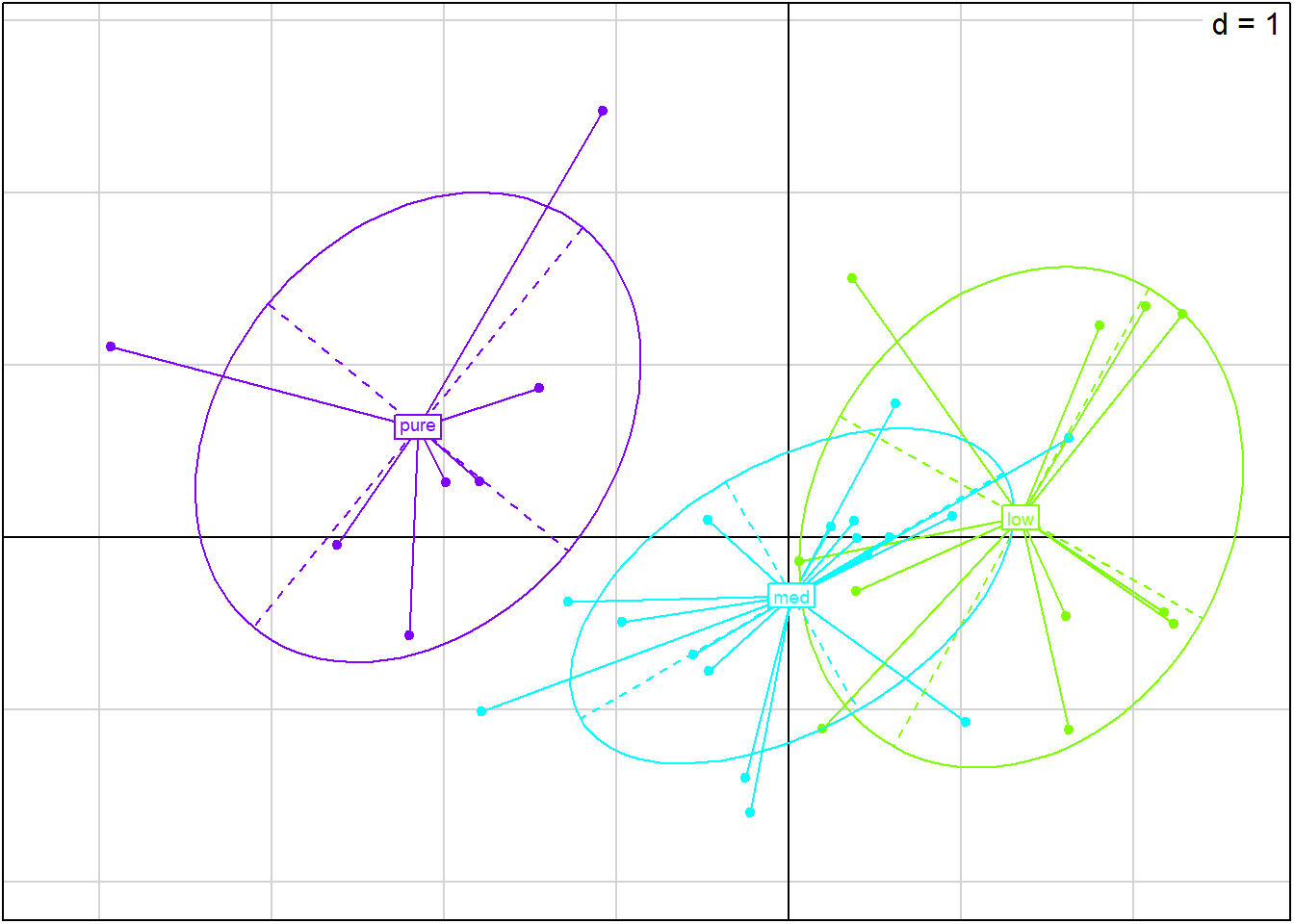
plot(acp, gg = T, cex.row = .6, col.row = 4,
cex.col = .6, col.col = "darkgrey")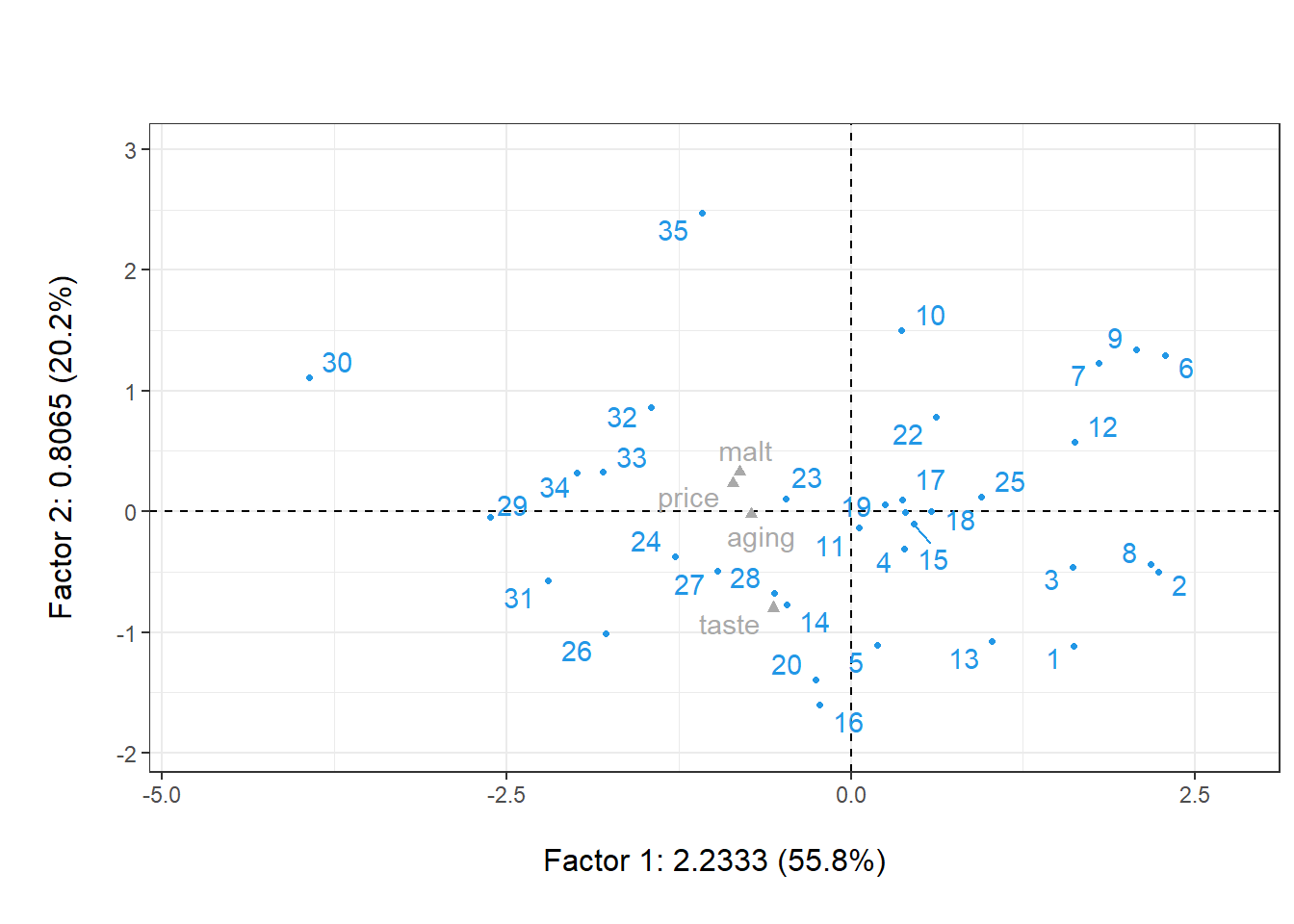
( sup <- supqual(acp, Whisky[, 3]) )$wcat
low med pure
0.3143 0.4857 0.2000
$dis2
low med pure
1.8445 0.1645 5.6113
$coor
Axis1 Axis2 Axis3 Axis4
low 1.34595 0.1174 -0.1214 0.06628
med 0.01583 -0.3391 -0.1630 0.15082
pure -2.15349 0.6389 0.5867 -0.47043
$tv
Axis1 Axis2 Axis3 Axis4
low 3.55539 0.516 -0.6041 0.4549
med 0.06001 -2.139 -1.1643 1.4861
pure -4.20128 2.074 2.1559 -2.3849
$cos2
Axis1 Axis2 Axis3 Axis4
low 0.982155 0.007472 0.007992 0.002382
med 0.001522 0.698702 0.161524 0.138251
pure 0.826463 0.072755 0.061343 0.039439
$scr
Axis1 Axis2 Axis3 Axis4
qual 1.497 0.1418 0.08638 0.056692.6 Taller (ACP de Lactantes)
if(!require(ISwR)){ # intenta cargar ISwR, si falla entonces
install.packages("ISwR") # instala ISwR
library(ISwR) # carga ISwR
}
data(kfm)
data <- kfm[, c(2,4:7)]
summary(data) dl.milk weight ml.suppl mat.weight mat.height
Min. : 4.44 Min. :4.12 Min. : 0.0 Min. :47.0 Min. :153
1st Qu.: 6.55 1st Qu.:4.98 1st Qu.: 16.2 1st Qu.:55.0 1st Qu.:162
Median : 7.66 Median :5.35 Median : 57.5 Median :58.0 Median :167
Mean : 7.50 Mean :5.32 Mean : 96.0 Mean :60.0 Mean :167
3rd Qu.: 8.43 3rd Qu.:5.64 3rd Qu.:103.8 3rd Qu.:64.5 3rd Qu.:172
Max. :10.43 Max. :6.58 Max. :590.0 Max. :80.0 Max. :185 summary(kfm[, 3]) boy girl
25 25 acp <- dudi.pca(data, scannf = FALSE, nf = n)inertia_acp <- inertia(acp, row.inertia = TRUE, col.inertia = TRUE)
valp <- t(inertia_acp$tot.inertia) # valores propios
kable(valp, digits = 2)| Ax1 | Ax2 | Ax3 | Ax4 | Ax5 | |
|---|---|---|---|---|---|
| inertia | 2.47 | 1.07 | 0.74 | 0.44 | 0.27 |
| cum | 2.47 | 3.54 | 4.28 | 4.73 | 5.00 |
| cum(%) | 49.49 | 70.90 | 85.70 | 94.59 | 100.00 |
kable(inertia_acp$col.abs, digits = 1)| Axis1 | Axis2 | Axis3 | Axis4 | Axis5 | |
|---|---|---|---|---|---|
| dl.milk | 27.4 | 2.4 | 15.7 | 19.3 | 35.2 |
| weight | 24.4 | 0.7 | 33.5 | 16.4 | 25.0 |
| ml.suppl | 0.3 | 90.0 | 0.0 | 1.3 | 8.4 |
| mat.weight | 23.0 | 4.1 | 27.6 | 35.4 | 10.0 |
| mat.height | 24.9 | 2.8 | 23.3 | 27.5 | 21.4 |
kable(inertia_acp$col.rel, digits = 1)| Axis1 | Axis2 | Axis3 | Axis4 | Axis5 | |
|---|---|---|---|---|---|
| dl.milk | -67.7 | -2.6 | 11.6 | 8.6 | -9.5 |
| weight | -60.4 | 0.7 | 24.8 | -7.3 | 6.8 |
| ml.suppl | -0.8 | 96.4 | 0.0 | -0.6 | -2.3 |
| mat.weight | -56.8 | -4.3 | -20.4 | -15.7 | -2.7 |
| mat.height | -61.7 | 3.0 | -17.2 | 12.2 | 5.8 |
s.corcircle(acp$co)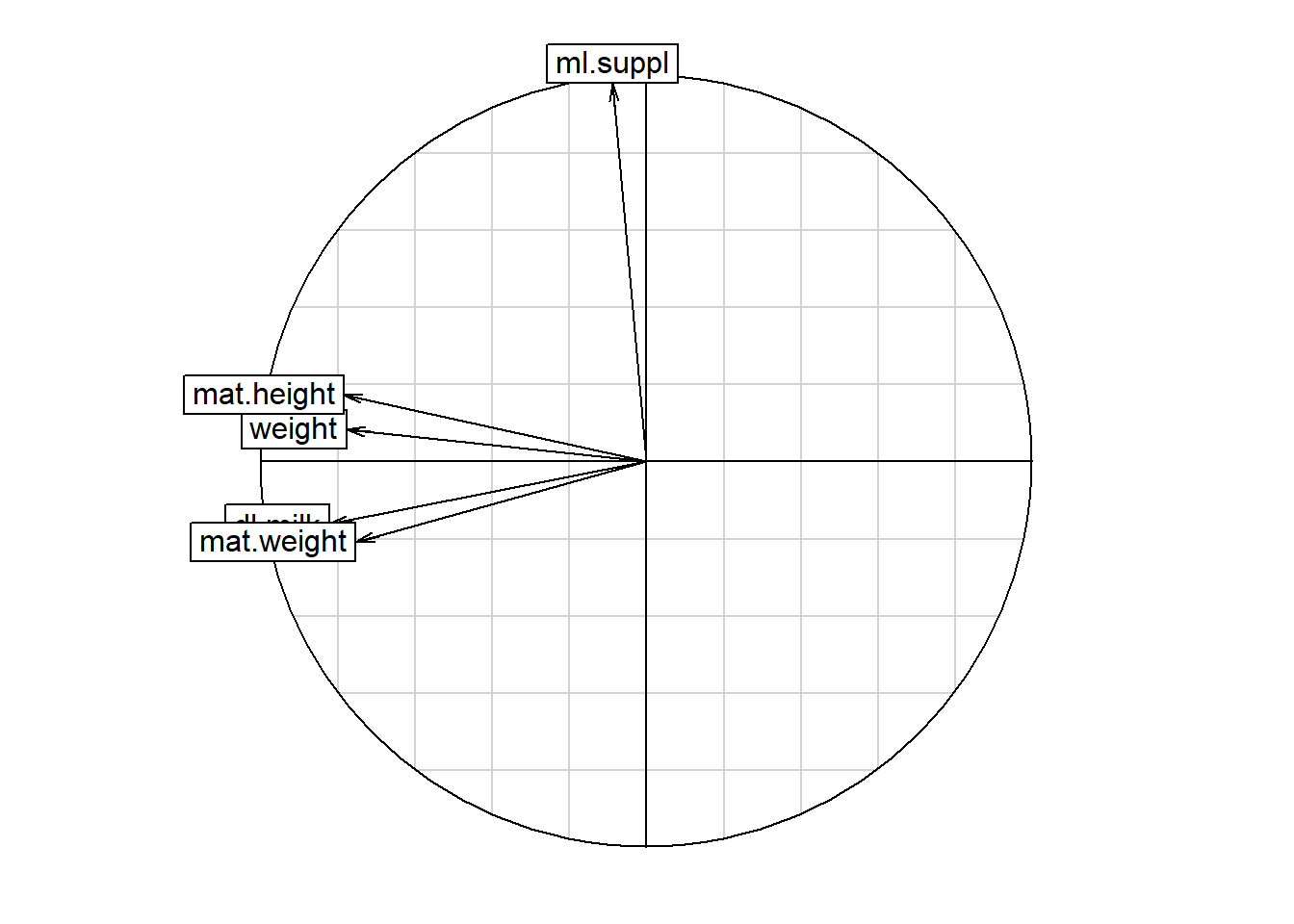
s.corcircle(acp$co, 3, 2)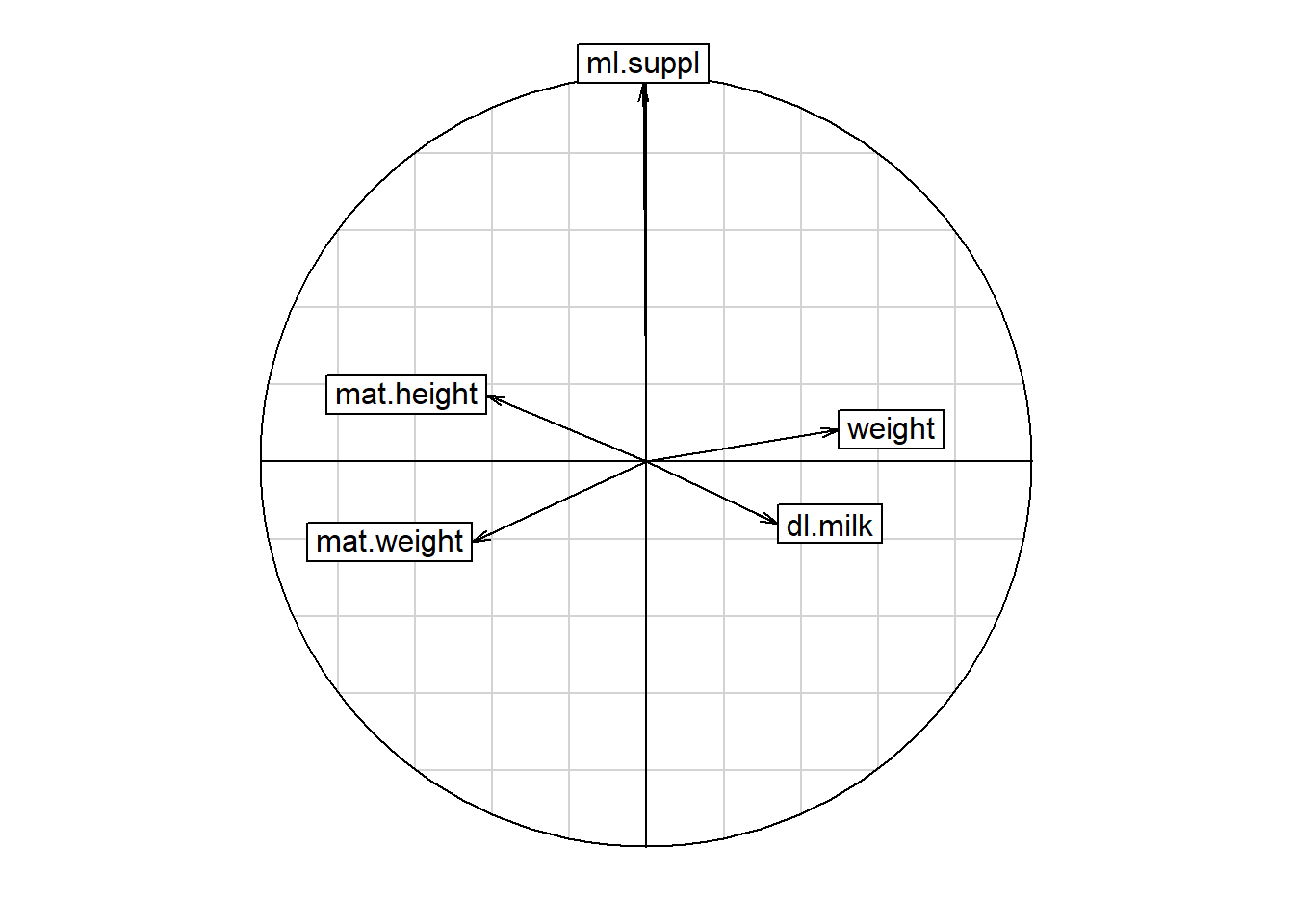
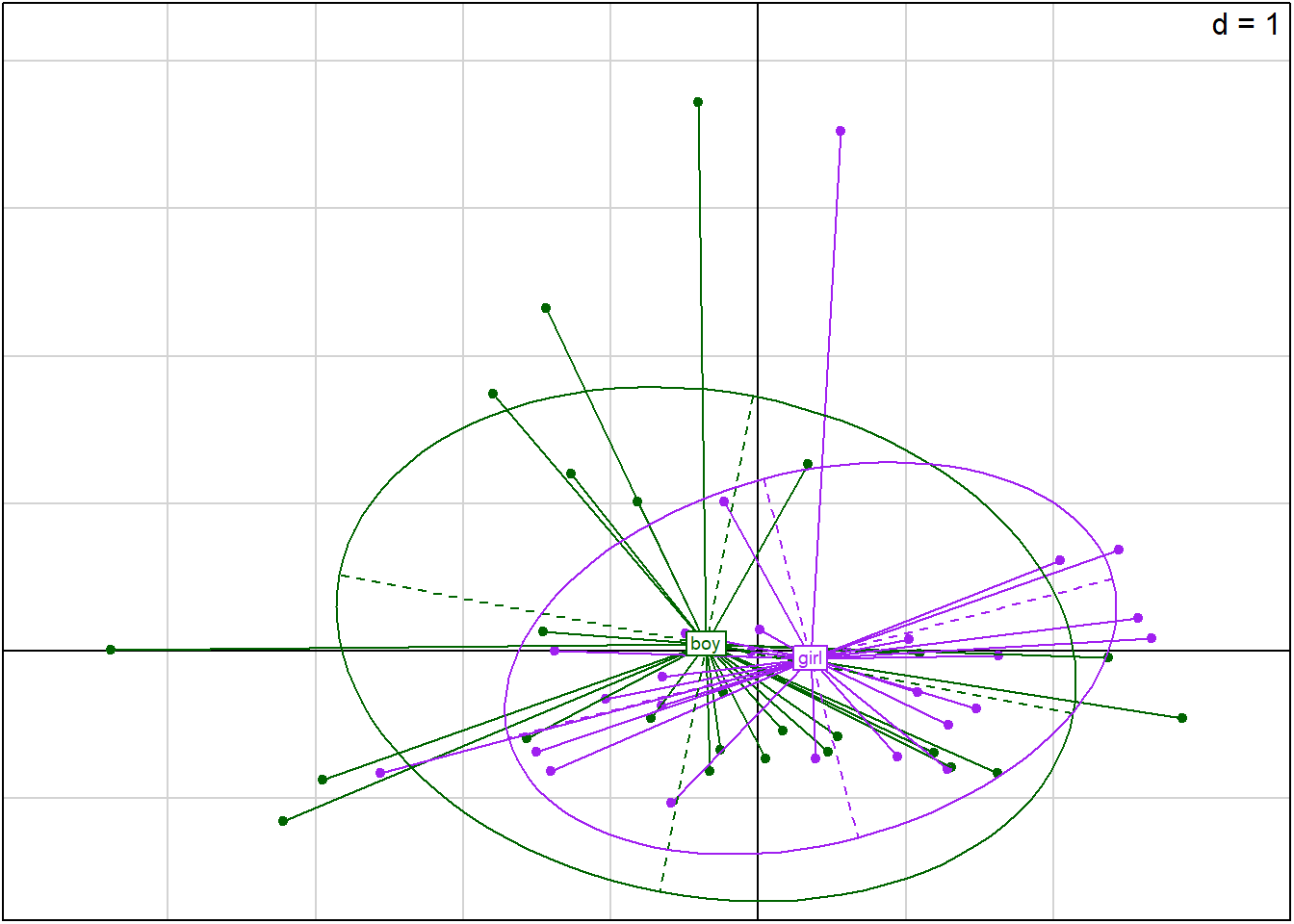
plot(acp, gg = T, cex.row = .5, col.row = 4,
Tcol = FALSE)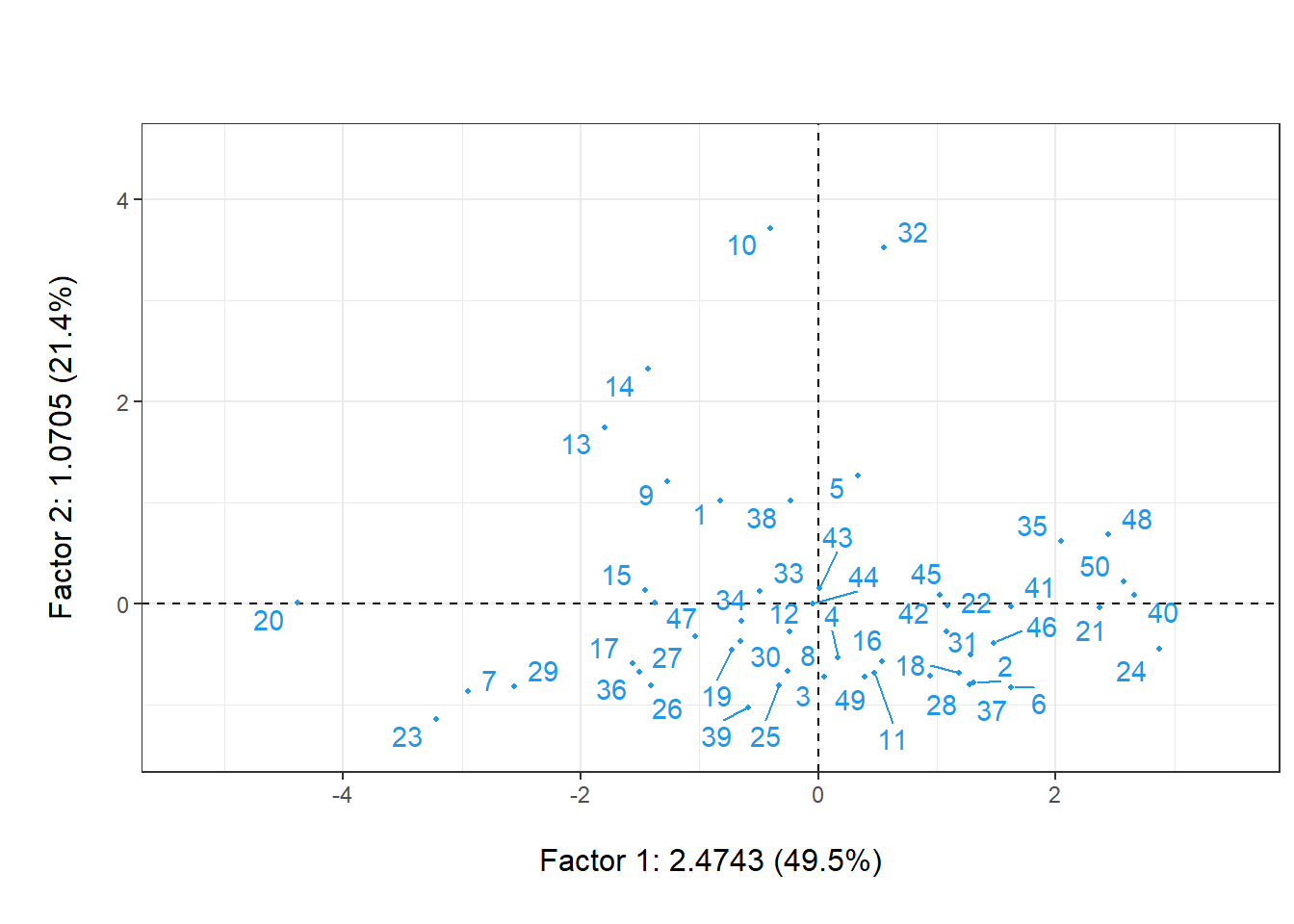
( sup <- supqual(acp, kfm[, 3]) )$wcat
boy girl
0.5 0.5
$dis2
boy girl
0.1598 0.1598
$coor
Axis1 Axis2 Axis3 Axis4 Axis5
boy -0.3536 0.04806 0.1604 0.06465 -0.0506
girl 0.3536 -0.04806 -0.1604 -0.06465 0.0506
$tv
Axis1 Axis2 Axis3 Axis4 Axis5
boy -1.574 0.3252 1.306 0.6788 -0.6809
girl 1.574 -0.3252 -1.306 -0.6788 0.6809
$cos2
Axis1 Axis2 Axis3 Axis4 Axis5
boy 0.7823 0.01445 0.1611 0.02616 0.01602
girl 0.7823 0.01445 0.1611 0.02616 0.01602
$scr
Axis1 Axis2 Axis3 Axis4 Axis5
qual 0.125 0.00231 0.02574 0.00418 0.00256